日本人集団を用いたメタボリックシンドローム
感受性遺伝子多型の解析
1 目次 第1 章 背景 ... 3 第1 節 内臓脂肪蓄積と異所性脂肪沈着 ... 3 第2 節 メタボリックシンドロームに関連する遺伝素因 ... 6 第2 章 日本人の疾患感受性 SNP を検討する意義 ... 9 第3 章 方法(Multi-plex PCR 法とインベーダー法) ... 11 第1 節 Multi-plex PCR 法... 11 第1 項 原理 ... 11 第2 項 プライマー設計 ... 11 第3 項 方法 ... 14 第2 節 インベーダー法 ... 17 第1 項 原理 ... 17 第2 項 方法 ... 17 第4 章 BMI・脂肪分布に関連した SNP の検討 ... 21 第1 節 サンプルと対象 SNP の遺伝子型決定 ... 24 第2 節 脂肪分布パラメーターや血清パラメーターとの関連解析 ... 27 第1 項 血清パラメーター ... 27 第2 項 統計解析 ... 30 第3 節 結果 ... 35 第1 項 BMI ... 35 第2 項 VFA ... 35 第3 項 SFA ... 36 第4 項 V/S 比 ... 36 第5 項 血清パラメーター ... 36 第4 節 考察 ... 37 第5 章 血中アディポネクチン量に関連した SNP の検討 ... 47 第1 節 サンプルと対象 SNP の遺伝子型決定 ... 49
2 第2 節 脂肪分布パラメーターと血清パラメーターの関連解析 ... 51 第1 項 アディポネクチン ... 51 第2 項 ヒトアディポネクチン ELISA キット(大塚製薬株式会社) ... 52 第3 項 統計解析 ... 57 第4 項 ハプロタイプ解析 ... 57 第3 節 結果 ... 58 第1 項 1st セットを用いた関連解析 ... 58 第2 項 2nd セットを用いた関連解析とメタ解析 ... 58 第3 項 血中アディポネクチン量との関連解析 ... 59 第4 項 ハプロタイプ解析 ... 59 第4 節 考察 ... 60 第6 章 異所性脂肪沈着(脂肪肝)に関連する SNP の検討 ... 71 第2 節 NAFLD との関連解析 ... 76 第3 節 脂肪分布パラメーターや血清パラメーターとの関連解析 ... 78 第1 項 血清パラメーター ... 78 第4 節 肝細胞の組織所見(重症度) ... 79 第5 節 各 SNP の独立した効果 ... 79 第6 節 結果 ... 80 第1 項 NAFLD との関連解析 ... 80 第2 項 脂肪分布パラメーターや血清パラメーターとの関連解析 ... 80 第3 項 肝細胞の組織所見(重症度)との関連解析 ... 80 第4 項 各 SNP の独立した効果 ... 81 第7 節 考察 ... 81 総括 ... 87 謝辞 ... 89 参考文献 ... 91
3
第
1 章 背景
第
1 節 内臓脂肪蓄積と異所性脂肪沈着
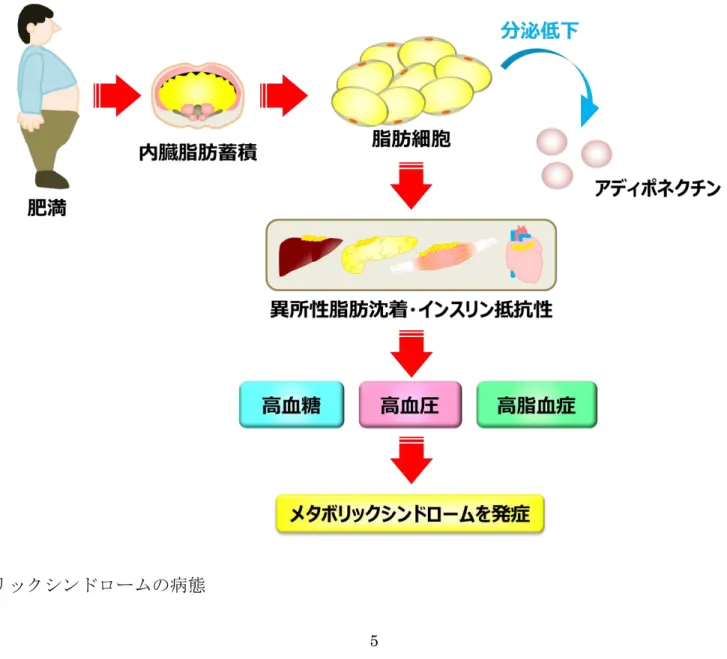
現在日本では、食生活の変化や運動不足によりメタボリックシンドロームが 増加してきており深刻な問題となっている。 メタボリックシンドロームの病態において主要な役割を担っているのは内臓 脂肪の過剰な蓄積である。さらに、内臓脂肪蓄積が限界量に達すると、蓄積し きれない余剰エネルギーは肝臓、骨格筋、膵臓、心臓などに蓄積し、異所性脂 肪沈着を起こすと考えられている(図1)[1]。 近年肥満の基礎研究が進み、肥満患者では脂肪組織由来の生理活性物質(ア ディポサイトカイン)の分泌異常を伴うことがわかってきた。異所性脂肪沈着 は、脂肪細胞から分泌されるアディポサイトカインの一つであるアディポネク チンによって燃焼されるべきものである。しかし、内臓脂肪が蓄積するとアデ ィポネクチンの分泌が低下し、炎症性のサイトカインの分泌が増加し、異所性 脂肪沈着(肝臓、膵臓、骨格筋、心臓等)とインスリン抵抗性を引き起こし、 その結果、高血糖や高血圧、高脂血症などの様々な異常を引き起こす。内臓脂 肪の蓄積とこれらのうち 2 つ以上を合併した状態をメタボリックシンドローム という(図2)。4 図 1 内臓脂肪蓄積と異所性脂肪沈着 [1]
5 図 2 メタボリックシンドロームの病態
6
第
2 節 メタボリックシンドロームに関連する遺伝素因
メタボリックシンドロームの発症の原因には食生活や運動などの環境要因も 重要であるが、遺伝素因も重要な役割を果たしていると考えられている。近年 のゲノムワイド関連解析(Genome Wide Association Study: GWAS)の進歩に よって体格指数(Body Mass Index: BMI)や脂肪分布などに関連する一塩基多 型(Single Nucleotide Polymorphism: SNP)が多数同定され、具体的な遺伝素
因が報告されてきている(表1)。 しかし、メタボリックシンドロームに関連するSNP の多くは、欧米人を対象 とした研究により見いだされたものである。SNP の頻度は人種、国、地域によ って異なり、日本人集団でも違いはわずかではあるが、本土クラスターと琉球 クラスターの2 つの主なクラスターに大別されることが報告されている(図 3) [90]。また、連鎖不平衡ブロックの構造も人種によって異なることが知られてい る(図4)[89]。日本人は、欧米人と比べると軽度な肥満でも様々な代謝異常を 起こしやすいことが知られており、これらのことから肥満や代謝異常に関わる SNP やその病態発症への寄与率には人種間で違いがあることが考えられる。
7
Japanese: 日本人 + Hondo: 本土 HapMap JPT: 東京在住の日本人 × Ryukyu: 琉球
HapMap CHB: 北京在住の中国漢民族 Other Japanese: 他の日本人 HapMap CEU: 西・北欧系ユタ州住民 HapMap JPT: 東京在住の日本人 HapMap YRI: ナイジュリアのヨルバ族 HapMap CHB: 北京在住の中国漢民族
8 図 4 人種による連鎖不平衡ブロックの違い
9
第
2 章 日本人の疾患感受性 SNP を検討する意義
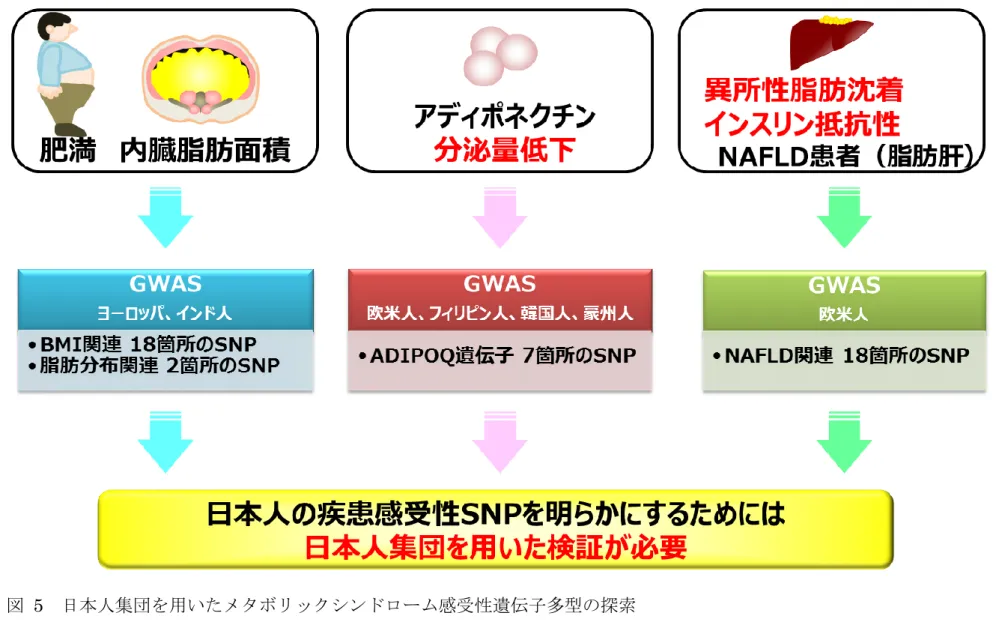
単一遺伝子疾患とは異なりメタボリックシンドロームのような多因子疾患は、 遺伝素因と環境要因が複雑に関わり合って発症することから、生活習慣が異な る欧米人の結果をそのまま日本人に当てはめることはできないと考えられる。 実際、欧米人で同定された疾患感受性SNP の中には、日本人では感受性が認め られないものが含まれている。したがって、日本人の疾患感受性SNP を明らか にするためには、日本人集団を用いた検証が必要である。 そこで本研究では、主に欧米人を対象に行われた GWAS 研究で報告された SNP のうち、BMI と脂肪分布、血中アディポネクチン量、異所性脂肪沈着とし て脂肪肝に関連するSNP に着目し、Multi-plex PCR 法とインベーダー法を用 いて遺伝子型を決定し、メタボリックシンドロームに関係する形質との関連性 を検討することで日本人における疾患感受性SNP を明らかにすることを目的と した(図5)。10
11
第
3 章 方法(Multi-plex PCR 法とインベーダー法)
第
1 節 Multi-plex PCR 法
第
1 項 原理
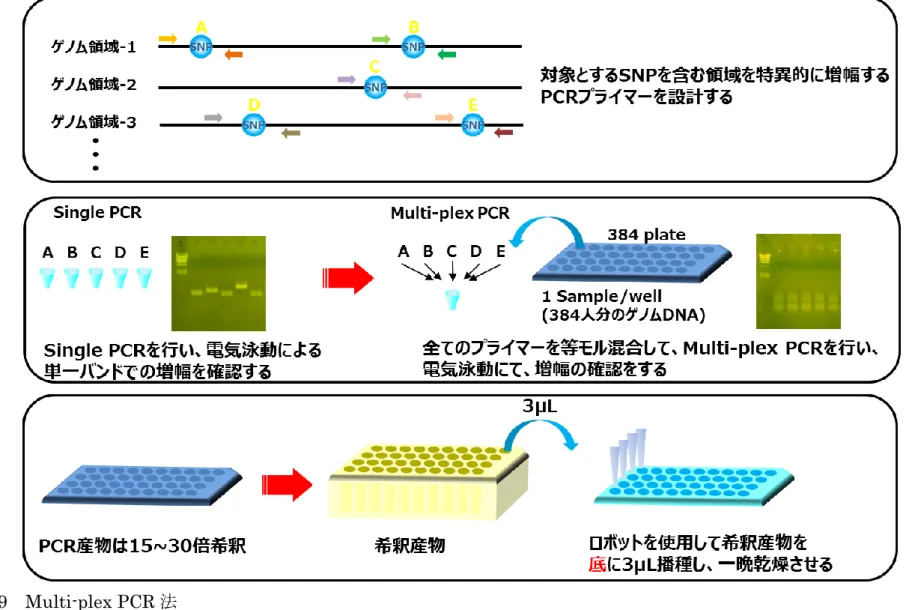
Multi-plex PCR 法は、対象とする SNP を含む領域を特異的に増幅する PCR プライマーをそれぞれのSNP に対して設計し、電気泳動で単一バンドでの増幅 を確認後、全てのプライマーを等モル混合してPCR を行うことで全ての対象領 域を一度に増幅する方法である。第
2 項 プライマー設計
① プライマーの配列 下記に示した 5 つの条件を全て含まないように設計を行い、増幅効率を高め るように考慮した。 A. 同一2 塩基の 3 回以上の繰り返し B. 任意の塩基の5 回以上の繰り返し C. プライマー末尾の3 塩基が同一の塩基 D. 同一3 塩基の 2 回以上の繰り返し E. 任意の塩基が1 塩基飛ばしで 4 回以上の繰り返し12 ② Tm 値 プライマー長は全て21 nt に固定した。 Tm 値は、Tm = 4℃ ×(G +C の合計数)+ 2℃ ×(A +T の合計数) の方法で計算を行い、対象となる全てのプライマーのTm 値が 58℃から 66℃の 範囲になるようにした。基本的には62℃以下になるように設計し、増幅効率を 揃えるように考慮した。 ③ 遺伝子多型 プライマー配列上に遺伝子多型(SNP、挿入・欠失)が存在することにより、 下記のようなことが生じることがある(図6~8)。 図 6 PCR 増幅の失敗
13 片側アレルのみの増幅が起こった場合には、ヘテロ接合体の遺伝子多型がホ モ接合体の結果となり、遺伝子型決定に誤りが生じる原因となる(図7)。 図 7 片側アレルのみの増幅 2 つのアレルの増幅効率が極端に異なる場合にも、ホモ接合体とヘテロ接合体 の分離がうまく出来ずに、遺伝子型決定に誤りが生じる原因となる。上図では がホモ接合体かヘテロ接合体なのか判定できない(図8)。 図 8 2 つのアレルの増幅効率の違い
14 そのため、NCBI の dbSNP(http://www.ncbi.nlm.nih.gov/snp/,build132) を参照し、プライマー配列上に遺伝子多型が存在しないようにすることで、PCR の成功率と遺伝子型決定の精度を高めるように考慮した。 ④ ホモロジー検索 プライマーの配列が、ゲノムDNA 上の対象 SNP を含む領域以外にも同じ配 列が存在した場合、非特異的な増幅が起こり、この場合にも遺伝子型決定の失 敗や誤りが生じる可能性がある。全く同じ配列ではなくてもプライマーの 3’側 がほとんど同じ場合には、遺伝子型決定の失敗や誤りが生じる可能性がある。 そ の た め 、 ヒ ト 全 ゲ ノ ム 配 列 ( UCSC hg19 : http://hgdownload.cse.ucsc.edu/downloads.html)を対象として、対象SNP を 含む領域以外には完全一致する配列が存在せず、ゲノムDNA 上の他のどの位置 に対しても必ず 2 塩基以上は異なる配列を選択した。その際に、プライマー配 列の 3’側半分(11 nt 以内)の位置が必ず 2 塩基以上異なるようにすることで PCR の成功率や遺伝子型決定の精度を高めるように考慮した。しかし、選べな い場合は1 塩基以上異なるように配列を選択した。
第
3 項 方法
Multi-plex PCR 法にて増幅する前に、対象 SNP を含む領域を特異的に増幅 出来ているかどうか確認するために、Single PCR 法(Test PCR)(表2、表 4) を行う必要がある。Single PCR 法を行った後、1% アガロースゲル(日本ジェ ネティクス株式会社)による電気泳動を行い、全てのプライマーセットが単一 バンドで増幅することを確認した。その後、全てのプライマーセットを等モル15
混合し、Multi-plex PCR 法(表 3、表 4)により増幅した(図 9)[2]。PCR 反
応の試薬組成と反応条件は以下の組成で行った。PCR 反応プレートは 384 well
PCR プレート(4titude)を使用した。シールは Adhesive PCR Seal(4titude) を使用した。 表 2 Single PCR (Test PCR) Genome (10 ng/μl) 1.4 μL 10× Vogelstein Buffer 1.4 μL 78 mM MgCl2 0.84 μL 25 mM dNTPs 0.84 μL 1 μM Primer_Forwared 1.4 μL 1 μM Primer_Reverse 1.4 μL
5 U/μL Taq polymerase (+Ab) 0.5 μL
Distilled water 6.22 μL
16 表 3 Multi-plex PCR Genome (10 ng/μl) 1.4 μL 10× Vogelstein Buffer 1.4 μL 78 mM MgCl2 0.84 μL 25 mM dNTPs 0.84 μL 100 μM Primer_Forwared 0.0125(×plex) μL 100 μM Primer_Reverse 0.0125(×plex) μL
5 U/μL Taq polymerase (+Ab) 0.5 μL
Distilled water Variable
Total 14 μL
25 mM dNTPs(タカラバイオ株式会社)
5 U/μL Taq polymerase(+Ab) (タカラバイオ株式会社) 100 μM Primer(シグマアルドリッチジャパン) 表4 PCR 反応条件 95℃ 5 min 1 cycle 95℃ 15 sec 60℃ 45 sec 40 cycles 72℃ 3 min 72℃ 3 min 1 cycle 4℃ ∞
17
第
2 節 インベーダー法
第
1 項 原理
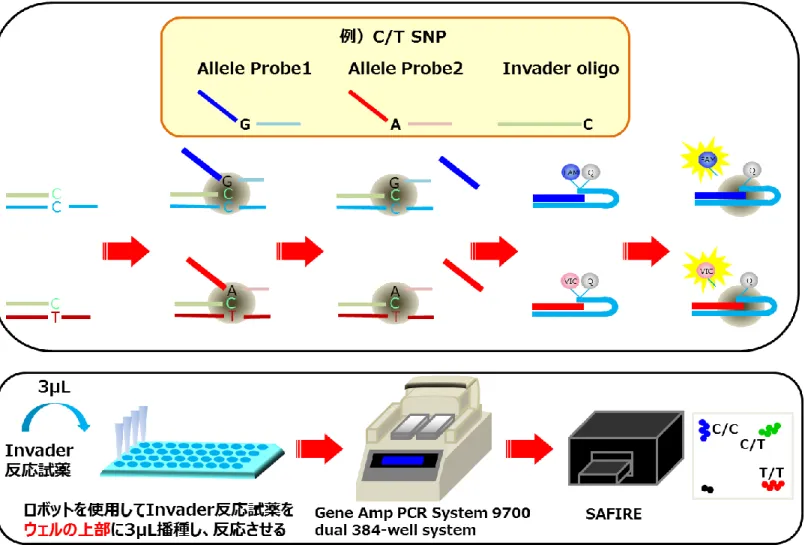
対象とするSNP に対して、蛍光色素配列(フラップ)を含むアレルプローブ 2 種類とインベーダープローブ 1 種類が必要である。 Multi-plex PCR 法による増幅産物に対して、アレルプローブとインベーダー プローブが、ハイブリダイゼーションすると 1 塩基でオーバーラップすること により、cleavase が認識する構造が形成される。cleavase がその構造を認識し て切断し、フラップを遊離させる。遊離したフラップが、検出用の DNA 基質 (FRET カセット)とハイブリダイズして新たな構造を形成する。この FRET カセットには蛍光色素(FAM と VIC)及び蛍光消光物質(Quencher)が修飾 されており、cleavase が、新たに形成された構造を認識して蛍光色素と蛍光消 光物質の間を切断する。蛍光色素が遊離され、蛍光を発光する。このサイクル が繰り返されることで蛍光シグナルが増幅される(図 10)。(Third WaveTechnologies, Madison, WI, USA)
参考ページ:http://hologic.co.jp/gene-cytology/invader/#linka
第
2 項 方法
Multi-plex PCR 法により増幅された PCR 産物を 15~30 倍希釈し、3 μL ずつ 384 well プレート(Sorenson)に播種した。一晩乾燥させ、インベーダー反応 試薬(蛍光値補正試薬ROX を含む)を各ウェル 3 μL ずつ播種し下記条件で反 応させた。SAFIRE(テカンジャパン株式会社)を用いて、FAM(励起波長 495 nm、蛍光波長 520 nm)、VIC(励起波長 526 nm、蛍光波長 550 nm)、ROX18
(励起波長580 nm、蛍光波長 610 nm)を測定し、FAM と VIC の値を ROX
の値でそれぞれ補正してプロットし、遺伝子型決定を行った(図10)。 インベーダー反応の試薬組成(表5)と反応条件(表 6)は以下の組成で行っ た。 表5 インベーダー反応の試薬組成 表6 インベーダー反応条件 95℃ 5 min 63℃ 15~60 min 20℃ ∞
GeneAmp 9700 PCR System (Life Technologies, Carlsbad, CA,USA)
Buffer 0.15 μL FRET 0.15 μL Cleavase 0.15 μL Probe mixture 0.15 μL Distilled water 2.4 μL Total 3 μL
19 図 9 Multi-plex PCR 法
20 図 10 インベーダー法
21
第
4 章 BMI・脂肪分布に関連した SNP の検討
近年、日本を含むアジア諸国では、食生活の変化や運動不足により肥満が増 加している。これはメタボリックシンドロームの主な特徴である。特に脂肪分 布の異常は、脂肪の絶対重量よりもメタボリックシンドロームにより密接に関 連している[3、4]。メタボリックシンドロームの病態において主要な役割を担っ ているのは内臓脂肪の過剰な蓄積である。内臓脂肪の過剰な蓄積が生じると、 アディポネクチンの分泌が低下し、異所性脂肪沈着とインスリン抵抗性を引き おこし、高血糖や高血圧、高脂血症などの異常を引き起こす。その結果、メタ ボリックシンドロームを発症することが明らかにされてきている[4、5]。 体脂肪分布は、ウエスト周囲長、ウェスト・ヒップ比、生体インピーダンス 法、またコンピュータ断層撮影(Computed Tomography: CT)によって測定さ れた内臓脂肪面積(Visceral Fat Area:VFA)や皮下脂肪面積(Subcutaneous Fat Area:SFA)によって求められる[3、10、11]。これらの体脂肪分布が遺伝 素因により影響されることがこれまでの研究結果から明らかにされてきた[6- 9]。 欧米人を対象としたGWAS により、ウエスト周囲長あるいは、ウエスト・ヒ ップ比に関連した SNP、CT により測定された内臓脂肪面積と皮下脂肪面積に 関連した SNP、肥満に関連した SNP が報告されている[12-15、19-21]。日本人においてはFTO (fat mass- and obesity-associate)遺伝子が皮下脂肪面
積に有意に関連することが当研究室により報告されている。また我々は、 LYPLAL1 (lysophospholipase-like protein 1)遺伝子の rs4846567 が内臓脂 肪面積:皮下脂肪面積比に関連していることや、SH2B1 (SH2B adaptor protein 1)遺伝子の rs7498665 が内臓脂肪面積に関連していることを報告して
22 きた[16-18、22]。 GWAS の進歩により肥満に感受性の遺伝子座に関する報告が増加しており、 最近新たにBMI に関連する 18 箇所の SNP や、脂肪分布に関連する 2 箇所の SNP が報告された(表 1)[23、24]。これらの SNP はヨーロッパ人・インド人 を対象とした解析で感受性が見出されたものである。そこで、これらの新たに 報告されたBMI あるいは脂肪分布関連 SNP について、日本人症例を用いて内 臓脂肪面積、皮下脂肪面積、内臓脂肪面積:皮下脂肪面積比との関連を検討し た。
23 表 1 GWAS で同定された BMI や脂肪分布などに関連する一塩基多型(SNP) BMI と関連があると報告されている遺伝子多型 rs1558902 FTO rs2867125 TMEM18 rs571312 MC4R rs10938397 GNPDA2 rs10767664 BDNF rs2815752 NEGR1 rs7359397 SH2B1 rs9816226 ETV5 rs3817334 MTCH2 rs29941 KCTD15 ウエストと体重に関連があると報告されている遺伝子多型 rs543874 SEC16B rs987237 TFAP2B rs7138803 FAIM2 rs10150332 NRXN3 新たに報告されたBMI と関連がある遺伝子多型 rs713586 ADCY3 rs12444979 GPRC5B rs2241423 MAP2K5 rs2287019 QPCTL rs1514175 TNNI3K rs13107325 SLC39A8 rs2112347 POC5 rs10968576 LINGO2 rs3810291 ZC3H4 rs887912 FLJ30838 rs13078807 CADM2 rs11847697 PRKD1 rs2890652 LRP1B rs1555543 PTBP2 rs4771122 MTIF3 rs4836133 ZNF608 rs4929949 STK33 rs206936 NUDT3 新たに報告された脂肪分布と関連がある遺伝子多型 rs2943650 IRS1 rs534870 SPRY2
24
第
1 節 サンプルと対象 SNP の遺伝子型決定
サンプルは、2002 年から 2011 年に外来を訪れた日本人集団 1424 人(男性 635 人、女性 789 人)を用いた。肥満は、日本の肥満の基準(BMI が 25 kg/m2 以上)[25]によって診断された(表 7)。インフォームドコンセントは各対象者 から書面で得ている。実施計画は、各研究所と京都大学の倫理委員会により承 認された(倫理委員会承認番号:G374)。 SNP は、ヨーロッパ人を対象として行われたメタ解析によって見出された BMI に関連する 18 箇所の SNP(rs1514175、rs1555543、rs713586、rs2112347、 rs206936、rs10968576, rs4929949、rs4771122、rs2241423、rs2287019、 rs3810291、rs2890652、rs887912、rs13078807、rs13107325、rs11847697、 rs12444979、rs4836133)と、ヨーロッパ人とインド人を対象として行われた メタ解析によって見出された脂肪分布に関連する2 箇所の SNP(rs2943650、 rs534870)の合計 20 箇所の SNP を対象とした(表 8)。1424 人の血液から抽出したゲノムDNA(Genomix kit, Talent Srl, Trieste,Italy)を鋳型として、そ
れぞれのSNP を含む領域を特異的に増幅する PCR プライマーを設計し増幅し
た。インベーダープローブを設計し(Third Wave Technologies, Madison, WI, USA)、インベーダー法にてそれぞれの SNP の遺伝子型決定を行った。
25 表7 対象者の臨床情報 男性 女性 全体 サンプル数 635 789 1424 年齢 (歳) 48.6 ± 12.5 52.3 ± 11.3 50.7 ± 12.0 体格指数BMI (kg/m2) 29.9 ± 6.0 28.2 ± 5.2 29.0 ± 5.6 内臓脂肪面積VFA (cm2) 153.9 ± 66.6 102.6 ± 54.1 125.5 ± 65.1 皮下脂肪面積SFA (cm2) 205.5 ± 108.3 243.3 ± 97.7 226.5 ± 104.3 血糖値 (mg/dL) 109.4 ± 31.9 108.4 ± 36.5 108.9 ± 34.5 インスリン (μU/mL) a 13.6 ± 17.9 10.4 ± 10.3 11.8 ± 14.3 インスリン抵抗性指数 a 3.9 ± 7.5 2.9 ± 3.8 3.4 ± 5.8 総 コ レ ス テ ロ ー ル (mg/dL) 211.4 ± 37.1 220.1 ± 38.6 216.2 ± 38.2 中性脂肪 (mg/dL) 170.0 ± 147.9 120.6 ± 80.8 142.6 ± 118.1 HDL- コ レ ス テ ロ ー ル (mg/dL) 51.7 ± 13.9 61.0 ± 16.0 56.8 ± 15.8 収縮期血圧(mmHg) 131.4 ± 16.8 130.5 ± 18.4 130.9 ± 17.8 拡張期血圧(mmHg) 84.7 ± 12.5 80.7 ± 11.2 82.4 ± 12.0
インスリン抵抗性指数(Homeostasis model assessment-Insulin Resistance: HOMA-IR) = 空腹時インスリン(U/mL) × 空腹時血糖値 (mg/dL) / 405 データは平均値±標準偏差
26 表8 対象 SNP 情報 関連性 SNP ID 染色体 番号 ポジション (GRCh37) 近傍の遺伝子名 BMI rs1514175 1 74,991,644 TNNI3K (TNNI3 interacting kinase)
BMI rs1555543 1 96,944,797 PTBP2 (polypyrimidine tract binding protein2) BMI rs713586 2 25,158,008 ADCY3 (adenylate cyclase 3)
BMI rs887912 2 59,302,877 FLJ30838
BMI rs2890652 2 142,959,931 LRP1B (low density lipoprotein receptor-related protein 1B ) 脂肪分布 rs2943650 2 227,105,921 IRS1 (insulin receptor substrate 1)
BMI rs13078807 3 85,884,150 CADM2 (cell adhesion molecule 2)
BMI rs13107325 4 103,188,709 SLC39A8 (solute carrier family 39 (zinc transporter), member 8) BMI rs2112347 5 75,015,242 POC5 (POC5 centriolar protein homolog)
BMI rs4836133 5 124,332,103 ZNF608 (Zinc Finger Protein 608)
BMI rs206936 6 34,302,869 NUDT3 (nudix (nucleoside diphosphate linked moiety X)-type motif 3) BMI rs10968576 9 28,414,339 LINGO2 (leucine rich repeat and Ig domain containing 2)
BMI rs4929949 11 8,604,593 STK33 (serine/threonine kinase 33)
BMI rs4771122 13 28,020,180 MTIF3 (mitochondrial translational initiation factor 3) 脂肪分布 rs534870 13 80,959,207 SPRY2 (sprouty homolog 2)
BMI rs11847697 14 30,515,112 PRKD1 (protein kinase D1)
BMI rs2241423 15 68,086,838 MAP2K5 (mitogen-activated protein kinase kinase 5)
BMI rs12444979 16 19,933,600 GPRC5B (G-protein coupled receptor family C group 5 member B) BMI rs2287019 19 46,202,172 QPCTL (glutaminyl-peptide cyclotransferase-like)
27
第
2 節 脂肪分布パラメーターや血清パラメーターとの関連解析
1424 人の内臓脂肪面積(VFA)と皮下脂肪面積(SFA)は CT [26]による測 定値を用いた。内臓脂肪面積と皮下脂肪面積の比(V/S 比)は VFA/ SFA として 解析した。内臓脂肪型肥満は、代謝障害に対する主要なリスク因子であるため[11、 30、31]、血清パラメーターとの関連性を解析した。 脂肪分布パラメーター(BMI、VFA、SFA、V/S 比)と血清パラメーター(血 糖値、インスリン、インスリン抵抗性指数、総コレステロール、中性脂肪、HDL コレステロール、収縮期血圧、拡張期血圧)を従属変数とし、遺伝子型(リス クアレル保有数により 0、1、2 と変換)、性別、年齢、BMI を説明変数として 多重線形回帰分析を行った。第
1 項 血清パラメーター
① 血糖値 [27] 血糖値は食事を摂取すると上昇するため、一般的には空腹時で検査する。正 常値は70~110 mg/dL 前後で、126 mg/dL 以上が糖尿病型とされる。 食後30 分位から血糖値は上昇し、1 時間後位にピークとなる。膵臓のランゲ ルハンス島(β細胞)から分泌されたインスリンよって血糖値は降下し、3 時間 後には空腹時レベルに戻る。糖は筋肉や肝臓に貯蔵される。 ② インスリン、インスリン抵抗性指数 [27] インスリンはランゲルハンス島(β細胞)から分泌され、血糖を抑制する作28 用を有する。 正常値は2~10 μU/mL であり、空腹時の血中インスリン値が 15 μU/mL 以上 を示す場合は、インスリン抵抗性の存在が考えられる。 糖尿病の患者ではインスリンを注射しても血糖値が下がりにくく、インスリ ンが効きにくい状態となる(インスリン抵抗性)。
インスリン抵抗性指数 HOMA-IR(Homeostasis model assessment-Insulin
Resistance)は、インスリン抵抗性の有無を簡便に調べるための指標の 1 つで、 下記の計算式で求められる。正常値は1.6 以下、インスリン抵抗性は 2.5 以上で ある。 HOMA-IR = 空腹時インスリン値(μU/mL)× 空腹時血糖値(mg/dL)÷ 405 ③ 総コレステロール、中性脂肪、HDL コレステロール [27] コレステロールは脂質(脂肪)の一種で、脂肪酸と結合したエステル型と、 分離した遊離型があり、これら二つを合わせて総コレステロール(T-Cho)とい う。正常値は130~199 mg/dL である。 中性脂肪は脂肪酸のグリセリンエステルのことで、血液中に含まれる中性脂 肪のほとんどはトリグリセリドである。本研究でも、中性脂肪はトリグリセリ ド値を使用した。動物における中性脂肪の主成分は飽和脂肪酸であり、血液中 の中性脂肪やコレステロールが増加しすぎる(脂質異常症)と、動脈硬化の危 険が高まる。中性脂肪の基準値は50~149 mg/dL である。 コレステロールを多く含んでいるリポタンパクとしてはHDL と LDL があり、 LDL コレステロールが増加すると活性酸素によって酸化され、酸化 LDL コレ ステロールは血管内皮へ接着し、血管壁内へ侵入する。酸化LDL コレステロー
29 ルは、マクロファージ(貪食細胞)によって食べ尽くされた後、マクロファー ジは泡沫細胞となる。修復のために血小板が集まり、粥腫(じゅくしゅ)を形 成する。このように血管を詰まらせ動脈硬化を引き起こす。これが脳動脈の場 合は脳梗塞に、心臓の冠状動脈の場合は心筋梗塞になる。 LDL コレステロールの正常値は 60~140 mg/dL である。一方、HDL コレス テロールは、血管に付着したコレステロールを剥がして、肝臓まで搬送し体外 に排出させる機能をもつ。正常値は40~65 mg/dL である。 <総コレステロール> 高値:脂質異常症(高脂血症)、動脈硬化、糖尿病、脂肪肝、閉塞性黄疸など 低値:肝硬変、肝臓癌、栄養障害など ④ 収縮期血圧、拡張期血圧 [27] 心臓は収縮と拡張を繰り返し活動している。心臓が収縮した時の最も高い血 圧が収縮期血圧であり、血液を心臓の中に溜めている間の最も低い圧力が拡張 期血圧である。正常値は収縮期血圧130 未満、拡張期血圧 85 未満の両者を満た す場合である。注意しなければならないのは、収縮期血圧 160、拡張期血圧 90 の場合である。脳梗塞や心筋梗塞を起こす危険性が高まるため、血圧を下げる ような指導や治療が必要である。
30
第
2 項 統計解析
ハーディーワインバーグ平衡は2テストで評価した[28]。多重線形回帰分析は R software (https://www.r-project.org/)を使用し、P 値はボンフェローニの 補正を使用して最も厳しく評価し、0.00096 (0.05/13 箇所の SNP/4 形質)未 満の値を統計学的に有意であるとした。また、脂肪分布は性的特異性があるこ とが知られているため[29,53]、男女別に解析を行った。 ① ハーディーワインバーグ平衡の適合度検定 ハーディーワインバーグ平衡の適合度検定は、集団の階層化や近親交配等の 確認に用いられる。一般的にピアソンの χ2適合度検定を用いて、観察された遺 伝子型の頻度と、ハーディーワインバーグ平衡が成立していると仮定した場合 の遺伝子型頻度の期待値を用いて検定を行う(表9)。 表9:χ2適合度検定の例 例)観察(Obs)された遺伝子型頻度 Allele 1 Both Allele 2 Total150 377 262 789
アレル頻度p1 = { 2×Obs (Allele 1) + Obs (Both) } / (2×Total) = (2×150 + 377) / (2×789)
= 0.42902
アレル頻度p2 = { Obs (Both) + 2×Obs (Allele 2) } / (2×Total) = (377 + 2×262 )/ (2×789)
31
ハーディーワインバーグ平衡の場合の期待値(Exp)は Exp (Allele 1) = (p1)2×Total = 0.42902×0.42902×789 = 145.2
Exp (Both) = 2×p1×p2×Total = 2×0.42902×0.57098×789 = 386.6 Exp (Allele 2) = (p2)2×Total = 0.57098×0.57098×789 = 257.2
χ2適合度検定
Obs: O、Exp: E、Allele 1: Al1、Both: B、Allele 2: Al2 χ2 = Σ (O - E)2 / E
= {(OAl1 - EAl1)2 / EAl1} + {(OB - EB)2 / EB} + {(OAl2 - EAl2)2 / EAl2}
= {(150 - 145.2)2 / 145.2} + {(377 - 386.6)2 / 386.6} + {(262 - 257.2)2 / 257.2} = 0.16 + 0.24 + 0.09 =0.49 帰無仮説:「ハーディーワインバーグ平衡に従っている」 対立仮説:「ハーディーワインバーグ平衡に従っていない」 自由度1(遺伝子型数 - アレルの数)で、危険率 5%となるのは χ2値が3.84 のときである。χ2値はそれ以下であることから、「ハーディーワインバーグ平衡 に従っている」という帰無仮説は棄却されない。 χ2値 < 3.84 : 帰無仮説が採択される χ2値 > 3.84 : 帰無仮説が棄却される 危険率5%のもとで有意な差があった場合は、帰無仮説を棄却して対立仮説を 採択することになるが、P 値は 0.48 であるため帰無仮説は棄却されない。
32 ② ハーディーワインバーグ平衡の適合度検定による実験精度の確認 図11 インベーダー法による遺伝子型決定の例 グレーの丸の部分( )の遺伝子型を誤って決定することにより、ハーディ ーワインバーグ平衡から逸脱してしまう場合がある(図11)。 ハーディーワインバーグ平衡から逸脱する理由は以下が考えられる。 A. 近傍に SNP や insertion、deletion が存在する B. 目的の領域とホモロジーが高い領域が存在する C. 増幅効率が極端に悪い よって、遺伝子型決定後にχ2適合度検定を行い評価した。 ③ 多重線形回帰分析 脂肪分布パラメーターや血清パラメーターを従属変数とし、遺伝子型(リス クアレル保有数により 0、1、2 と変換)、性別、年齢、BMI を説明変数として 多重線形回帰分析を行う場合、遺伝子型はAdditive Model を使用した。 Additive Model は片方のアレルの所有本数に比例してリスクが発生するとい
う関連の見方で、A/G の SNP でリスクアレルは G の場合は、A/A は 0、A/G は
33 多重線形回帰分析は下記の例の場合、内臓脂肪面積は、遺伝子型・年齢・BMI に関連するのか解析することが出来る(図12)。 図12 多重線形回帰分析の例 説明変数が 1 つの場合(内臓脂肪面積と遺伝子型との関連)は、回帰方程式 は直線になる。 図13 説明変数が 1 つの場合
34 説明変数が 2 つの場合(内臓脂肪面積と遺伝子型・年齢との関連)は、回帰 方程式は平面になる。 図14 説明変数が 2 つの場合 説明変数が3 つ以上の場合(内臓脂肪面積と遺伝子型・年齢・BMI との関連) は、R software を使用し、解析を行う。 ④ ボンフェローニ(Bonferroni)の補正 検定全体の有意水準を検定数で割った値を有意水準とする。補正が厳しくか かりすぎる面がある。 例)5 箇所の SNP で検定した場合 0.05 / 5 = 0.01 0.01 未満の値が統計学的に有意であるとなる。
35
第
3 節 結果
20 箇所の SNP のインベーダー法による遺伝子型決定の成功率は 99.0%以上 であった。ヨーロッパ人やインド人とは異なり、日本人集団では6 箇所の SNP (rs2890652、rs887912、rs13078807、rs13107325、rs11847697、rs12444979) はmonomorphic であり、rs4836133 は triallelic であったためこれらの SNP を 以降の解析から除外し、最終的には13 箇所の SNP を解析対象とした。遺伝子 型決定の結果はTable にまとめた通りである(表 10)。第
1 項 BMI
rs206936(NUDT3 遺伝子)の G アレルが女性においてのみ BMI の増加(P = 5.3×10-5)に有意な関連を示した。この関連は男性では認められなかった(P = 0.19)。rs1555543(PTBP2 遺伝子)は男性で BMI の増加(P = 0.017)に弱い 関連を示したが、有意ではなかった(表11)。第
2 項 VFA
13 箇所の SNP 全てにおいて、VFA とは有意な関連がみられなかった。 rs2112347(POC5 遺伝子)(P = 0.032)と rs206936(NUDT3 遺伝子)(P = 0.018) が女性で VFA の増加に弱い関連を示したが BMI で補正すると弱い関連も消失 した。rs2943650(IRS1 遺伝子)は女性で VFA の増加(P = 0.041)に弱い関 連(BMI で補正)を示した(表 12)。36
第
3 項 SFA
rs206936(NUDT3 遺伝子)の G アレルが女性においてのみ SFA の増加(P = 3.9×10-4)と有意な関連を示した。この関連についても男性では認められなか った(P = 0.10)。BMI と同様の結果が得られたが、この関連は BMI で補正す ることにより消失した。rs4771122(MTIF3 遺伝子)は男性で SFA の増加(P = 0.027)に弱い関連を示したが有意ではなく、BMI で補正すると弱い関連も消失 した。rs3810291(ZC3H4 遺伝子)は女性で SFA の増加(P = 0.0083)に弱い 関連(BMI で補正)を示したが、有意ではなかった(表 13)。第
4 項 V/S 比
13 箇所の SNP 全てにおいて、V/S 比について関連を示す SNP はなかった(表 14)。第
5 項 血清パラメーター
13 箇所の SNP 全てにおいて、血清パラメーター(代謝障害)との有意な関 連はみられなかった(表15)。女性でBMI と SFA に有意な関連がみられた NUDT3 遺伝子の rs206936 G
アレルに関しても、どの代謝形質とも関連がみられなかった。
これは、デンマーク人(男性3,004 人、女性 3,035 人)を対象として NUDT3
37
第
4 節 考察
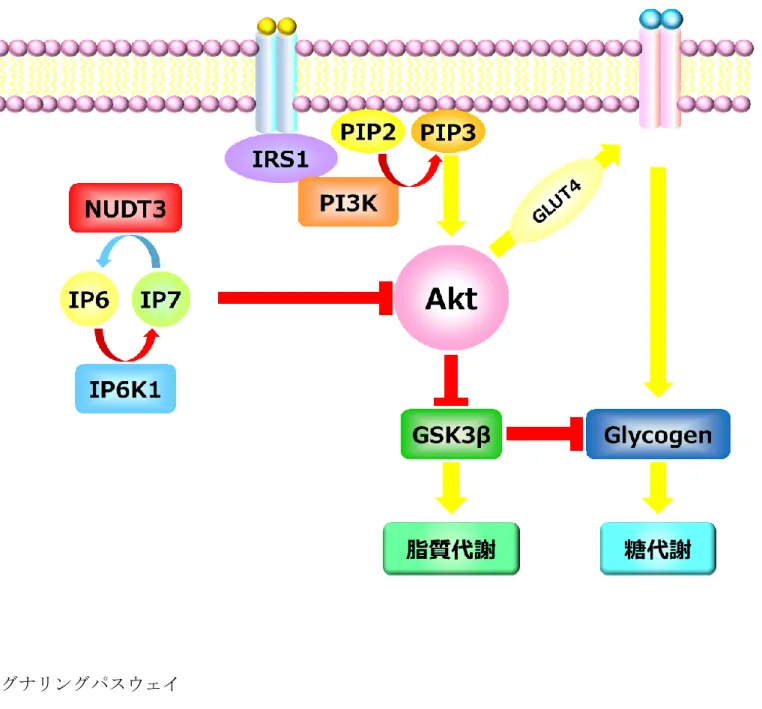
本研究では、最近の欧米人を対象としたGWAS により報告された肥満に関連 する13 箇所の SNP について、日本人症例を用いて関連解析を行った。脂肪分 布は性的特異性があることが知られているため、本研究では男女別に関連解析 を行った。その結果、女性においてのみrs206936(NUDT3 遺伝子)の G アレ ルに、BMI の増加(P = 5.3×10-5)やSFA の増加(P = 3.9×10-4)との有意な関 連がみられた。rs206936 で観察された SFA との関連は BMI で補正するとみら れなくなることから、BMI の増加に伴う皮下脂肪の蓄積に関与する SNP であ ると考えられた。 今後さらに多くの人数による解析や、男女別での解析による検証が必要であ ると考えられるが、本研究により rs206936 が日本人で BMI に感受性の SNP であることを見出すとともに、性差があることを示唆し、新たな知見を得た。 ヨーロッパ人のゲノムワイド関連メタ解析によると、NUDT3 遺伝子の rs206936 のリスクアレル頻度は 0.21 で、本研究で用いた日本人集団では 0.57 であったことから、人種間で頻度が大きく異なることが示唆された。 無機リン酸塩のジホスホイノシトール五リン酸(IP7)は Akt シグナリング[35] を介して、脂質代謝や糖代謝に関与することが知られている。NUDT3 は、ジホ スホイノシトール五リン酸(IP7)を脱リン酸化することから [33、34]、Akt シグナリングに何らかの影響を及ぼして、脂質代謝や糖代謝に関連することが 考えられた(図15)。本研究では、NUDT3 遺伝子の rs206936 と BMI や SFA との関連以外には有
意な関連はみられなかったが、rs2112347(POC5 遺伝子)(P = 0.032)と rs206936(NUDT3 遺伝子)(P = 0.018)が女性で VFA の増加と弱い関連がみ
38 に弱い関連を示す[15]という報告があることから、rs2112347(POC5 遺伝子) は内臓脂肪蓄積に関与している可能性が考えられた。 rs2943650(IRS1 遺伝子)は女性で VFA の増加(P = 0.041)に弱い関連を 示した。rs2943650(IRS1 遺伝子)は V/S 比の増加(P = 6.1×10-6)に関連を 示す[24]という報告があることから、rs2943650(IRS1 遺伝子)は脂肪分布に 関連している可能性も考えられた。 今後、これらのSNP をさらに多くの人数による解析を行うことで、日本人に おける肥満感受性SNP が明らかになるだろうと考えられる。
39 表10 対象者の遺伝子型決定結果、リスクアレル頻度、ハーディーワインバーグ平衡 SNP ID 近傍の遺 伝子名 アレル1/ アレル2 リスク アレル 遺伝子型 リスクアレル頻度 HWE P 値 男性 女性 男性 女性 男性 女性 11/12/22 11/12/22 rs1514175 TNNI3K A/G A 429/190/14 562/207/20 0.83 0.84 0.18 0.86 rs1555543 PTBP2 A/C C 16/162/453 13/209/566 0.85 0.85 0.74 0.20 rs713586 ADCY3 C/T C 154/337/142 243/368/178 0.51 0.54 0.10 0.088 rs2943650 IRS1 C/T T 9/121/503 11/119/659 0.89 0.91 0.58 0.040 rs2112347 POC5 G/T T 195/314/118 248/367/168 0.44 0.45 0.67 0.14 rs206936 NUDT3 A/G G 110/303/220 150/377/262 0.59 0.57 0.75 0.49 rs10968576 LINGO2 A/G G 392/209/28 519/240/28 0.21 0.19 0.98 0.97 rs4929949 STK33 C/T C 101/333/196 160/372/255 0.42 0.44 0.040 0.25 rs4771122 MTIF3 A/G G 375/229/30 446/302/41 0.23 0.24 0.51 0.27 rs534870 SPRY2 A/G A 186/329/117 256/399/134 0.55 0.58 0.18 0.31 rs2241423 MAP2K5 A/G G 245/299/87 310/370/108 0.37 0.37 0.78 0.89 rs2287019 QPCTL C/T C 425/186/22 499/258/30 0.82 0.80 0.77 0.64 rs3810291 ZC3H4 A/G A 52/219/360 47/269/471 0.26 0.23 0.026 0.30 HWE, ハーディーワインバーグ平衡(P 値 0.05 未満を太字) 11, アレル 1/アレル 1 12, アレル 1/アレル 2 22, アレル 2/アレル 2
40 表11 13 箇所の SNP と BMI との関連解析 SNP ID 近傍の 遺伝子名 男性 女性 遺伝子型による平均値 (kg/m2) リスクアレルに対する 効果 遺伝子型による平均値 (kg/m2) リスクアレルに対する 効果 11 12 22 β(標準誤差) P 値 11 12 22 β(標準誤差) P 値 rs1514175 TNNI3K 29.5 ± 5.3 30.9 ± 7.3 31.0 ± 4.9 -0.014 ( 0.006) 0.015 28.1 ± 5.0 28.3 ± 5.7 28.7 ± 4.8 -0.003 ( 0.005) 0.59 rs1555543 PTBP2 27.7 ± 4.2 29.4 ± 4.5 30.2 ± 6.4 0.014 ( 0.006) 0.017 31.2 ± 6.7 28.4 ± 5.8 28.0 ± 4.9 -0.009 ( 0.005) 0.095 rs713586 ADCY3 30.0 ± 6.0 30.1 ± 6.4 29.4 ± 4.9 0.001 ( 0.004) 0.77 28.1 ± 5.0 28.2 ± 5.2 28.4 ± 5.6 -0.002 ( 0.004) 0.66 rs2943650 IRS1 31.6 ± 5.3 30.1 ± 5.1 29.9 ± 6.2 -0.008 ( 0.007) 0.25 26.8 ± 3.9 28.4 ± 6.0 28.2 ± 5.1 0.002 ( 0.006) 0.78 rs2112347 POC5 30.1 ± 6.0 30.0 ± 5.8 29.2 ± 4.8 -0.005 ( 0.004) 0.24 28.1 ± 5.8 28.3 ± 4.8 28.3 ± 5.2 0.004 ( 0.004) 0.28 rs206936 NUDT3 30.2 ± 6.1 30.2 ± 6.3 29.4 ± 5.5 -0.006 ( 0.004) 0.19 27.1 ± 4.7 28.0 ± 5.1 29.0 ± 5.6 0.015 ( 0.004) 5.3×10-5 rs10968576 LINGO2 30.1 ± 6.2 29.6 ± 5.7 30.0 ± 4.3 -0.001 ( 0.005) 0.79 28.2 ± 5.3 28.2 ± 5.0 27.5 ± 5.3 -0.003 ( 0.005) 0.60 rs4929949 STK33 29.8 ± 4.9 29.9 ± 6.2 30.0 ± 6.2 0.001 ( 0.004) 0.76 28.3 ± 5.4 28.5 ± 5.3 27.7 ± 4.9 0.005 ( 0.004) 0.14 rs4771122 MTIF3 29.8 ± 5.8 30.2 ± 6.4 30.4 ± 4.5 0.007 ( 0.005) 0.16 28.2 ± 5.1 28.1 ± 5.1 29.1 ± 7.3 0.002 ( 0.004) 0.69 rs534870 SPRY2 29.8 ± 6.2 30.0 ± 6.1 29.9 ± 5.4 -0.002 ( 0.004) 0.62 28.0 ± 5.0 28.3 ± 5.3 28.4 ± 5.3 -0.003 ( 0.004) 0.51 rs2241423 MAP2K5 29.7 ± 5.6 29.9 ± 6.1 30.7 ± 6.6 0.005 ( 0.004) 0.24 27.7 ± 5.0 28.6 ± 5.2 28.4 ± 5.8 0.006 ( 0.004) 0.13 rs2287019 QPCTL 30.0 ± 6.2 29.7 ± 5.5 30.1 ± 4.8 0.001 ( 0.005) 0.92 28.1 ± 5.1 28.3 ± 5.3 27.4 ± 4.5 -0.001 ( 0.005) 0.82 rs3810291 ZC3H4 31.1 ± 7.4 30.1 ± 6.2 29.6 ± 5.6 0.007 ( 0.005) 0.13 26.9 ± 3.4 28.3 ± 5.4 28.2 ± 5.2 -0.004 ( 0.004) 0.32 BMI は平均値±標準偏差で示し、P 値は年齢で補正した値を示した。 11, アレル 1/アレル 1 12, アレル 1/アレル 2 22, アレル 2/アレル 2
41 表12 13 箇所の SNP と VFA との関連解析 SNP ID 近傍の 遺伝子名 男性 女性 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 11 12 22 β(標準誤差) P 値 P 値* 11 12 22 β(標準誤差) P 値 P 値* rs1514175 TNNI3K 152.6 ± 68.9 157.1 ± 61.8 157.5 ± 59.7 -0.024 ( 0.016) 0.14 0.74 103.3 ± 53.7 101.8 ± 56.1 93.3 ± 44.2 0.013 ( 0.018) 0.45 0.16 rs1555543 PTBP2 130.9 ± 70.1 150.4 ± 66.4 155.8 ± 66.3 0.027 ( 0.016) 0.098 0.58 129.7 ± 60.9 104.9 ± 54.7 101.3 ± 53.6 -0.02 ( 0.018) 0.29 0.97 rs713586 ADCY3 160.6 ± 69.0 151.9 ± 63.2 151.8 ± 71.9 0.02 ( 0.012) 0.11 0.093 101.7 ± 49.6 98.3 ± 51.3 112.8 ± 63.6 -0.012 ( 0.013) 0.33 0.36 rs2943650 IRS1 148.5 ± 59.3 165.5 ± 75.7 151.2 ± 64.2 -0.029 ( 0.019) 0.13 0.27 92.9 ± 38.4 110.3 ± 57.0 101.4 ± 53.7 -0.032 ( 0.022) 0.15 0.041 rs2112347 POC5 159.6 ± 66.3 151.1 ± 67.0 152.0 ± 65.6 -0.014 ( 0.012) 0.26 0.52 96.9 ± 58.5 105.3 ± 50.8 104.2 ± 52.5 0.027 ( 0.013) 0.032 0.058 rs206936 NUDT3 155.2 ± 65.4 155.3 ± 67.7 151.3 ± 66.0 -0.008 ( 0.012) 0.53 0.99 99.9 ± 54.7 100.3 ± 55.3 107.6 ± 51.7 0.03 ( 0.013) 0.018 0.88 rs10968576 LINGO2 152.4 ± 66.5 157.2 ± 68.5 158.5 ± 58.5 0.014 ( 0.015) 0.34 0.22 102.4 ± 53.0 103.9 ± 55.8 93.7 ± 58.2 -0.004 ( 0.016) 0.83 0.89 rs4929949 STK33 163.1 ± 72.7 154.1 ± 64.1 149.8 ± 67.8 0.018 ( 0.013) 0.16 0.16 109.5 ± 58.6 102.4 ± 54.3 98.5 ± 50.1 0.018 ( 0.013) 0.16 0.50 rs4771122 MTIF3 152.9 ± 68.8 155.5 ± 62.8 153.4 ± 69.1 0.008 ( 0.014) 0.56 0.90 102.9 ± 54.6 101.8 ± 53.7 105.5 ± 51.7 0.004 ( 0.015) 0.80 0.98 rs534870 SPRY2 150.5 ± 60.5 155.1 ± 72.1 157.0 ± 59.8 -0.008 ( 0.012) 0.53 0.65 103.9 ± 55.6 103.0 ± 54.1 98.9 ± 51.2 0.008 ( 0.013) 0.55 0.19 rs2241423 MAP2K5 158.2 ± 72.5 152.2 ± 62.3 148.9 ± 64.7 -0.007 ( 0.012) 0.58 0.19 98.5 ± 52.6 105.8 ± 54.7 104.5 ± 55.5 0.024 ( 0.013) 0.07 0.26 rs2287019 QPCTL 153.5 ± 67.9 154.7 ± 65.6 160.1 ± 48.7 -0.013 ( 0.015) 0.41 0.31 100.8 ± 53.0 105.8 ± 56.0 101.0 ± 52.3 -0.006 ( 0.016) 0.69 0.74 rs3810291 ZC3H4 151.8 ± 65.4 153.7 ± 63.8 153.6 ± 68.2 0.002 ( 0.013) 0.85 0.52 101.4 ± 47.9 100.0 ± 55.0 104 .0± 54.0 -0.009 ( 0.015) 0.53 1.00 VFA は平均値±標準偏差で示し、P 値は年齢で補正した値、P 値*は年齢と BMI で補正した値を示した。 11, アレル 1/アレル 1 12, アレル 1/アレル 2 22, アレル 2/アレル 2
42 表13 13 箇所の SNP と SFA との関連解析 SNP ID 近傍の 遺伝子名 男性 女性 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 11 12 22 β(標準誤差) P 値 P 値* 11 12 22 β(標準誤差) P 値 P 値* rs1514175 TNNI3K 197.4 ± 102.7 221.5 ± 119.4 223.5 ± 101.3 -0.042 ( 0.016) 0.0089 0.24 243.1 ± 96.8 242.7 ± 100.1 254.2 ± 102.3 -0.002 ( 0.012) 0.85 0.80 rs1555543 PTBP2 172 ± 88.3 198 ± 95.3 208.6 ± 112.9 0.03 ( 0.016) 0.064 0.94 289.7 ± 86.8 247.4 ± 105 241 ± 94.9 -0.017 ( 0.012) 0.18 0.89 rs713586 ADCY3 209.5 ± 114.9 208.1 ± 108 194.4 ± 101.9 0.01 ( 0.012) 0.41 0.36 239.7 ± 92.4 245.2 ± 100.9 244.4 ± 98.6 -0.003 ( 0.008) 0.73 0.98 rs2943650 IRS1 266.9 ± 140.9 212.2 ± 101.7 202.8 ± 109.1 -0.036 ( 0.019) 0.052 0.099 227.5 ± 90.9 251.4 ± 100.6 242.1 ± 97.4 -0.01 ( 0.015) 0.48 0.16 rs2112347 POC5 208.6 ± 106.8 206.8 ± 110.8 194.4 ± 100.6 -0.016 ( 0.012) 0.18 0.49 240.5 ± 105.2 246.3 ± 93.4 242.9 ± 95.7 0.008 ( 0.008) 0.35 0.87 rs206936 NUDT3 216 ± 123 210.7 ± 106.3 193.1 ± 102.6 -0.019 ( 0.012) 0.1 0.32 222.9 ± 84.6 243 ± 98.1 255.4 ± 102.5 0.031 ( 0.009) 0.00039 0.42 rs10968576 LINGO2 208.3 ± 111.7 201.2 ± 103.8 205 ± 98.3 -0.003 ( 0.014) 0.85 0.98 241.5 ± 97.4 247.6 ± 98 241.4 ± 102.1 0.005 ( 0.011) 0.63 0.22 rs4929949 STK33 205.4 ± 101.6 206.8 ± 109.7 203.7 ± 110.3 0.009 ( 0.012) 0.46 0.45 244.3 ± 97.4 246.6 ± 101.1 238 ± 92.6 0.008 ( 0.009) 0.32 0.93 rs4771122 MTIF3 201.5 ± 108.8 207.2 ± 103.6 243.5 ± 133.5 0.031 ( 0.014) 0.027 0.075 242.9 ± 98.7 241.1 ± 95.7 264.1 ± 101.9 0.009 ( 0.01) 0.36 0.37 rs534870 SPRY2 208.8 ± 114.6 202.1 ± 109.5 208.7 ± 94.8 -0.009 ( 0.012) 0.47 0.59 237.9 ± 94.7 244.9 ± 98.6 249 ± 101.2 -0.009 ( 0.009) 0.33 0.53 rs2241423 MAP2K5 205.6 ± 106 201.8 ± 109 217.8 ± 114 0.004 ( 0.012) 0.73 0.40 243.3 ± 98.9 240.5 ± 94.6 253.7 ± 105.1 0.004 ( 0.009) 0.69 0.32 rs2287019 QPCTL 205.3 ± 105.9 202.4 ± 113.1 233.8 ± 115.9 -0.009 ( 0.015) 0.55 0.30 245.6 ± 100.9 240.2 ± 91.8 225.7 ± 95.1 0.007 ( 0.011) 0.50 0.20 rs3810291 ZC3H4 215 ± 123.9 207.1 ± 111.2 202.8 ± 104.2 0.004 ( 0.013) 0.77 0.19 239.7 ± 82.5 250.3 ± 98.5 240 ± 98.8 0.011 ( 0.01) 0.28 0.0083 SFA は平均値±標準偏差で示し、P 値は年齢で補正した値、P 値*は年齢と BMI で補正した値を示した。 11, アレル 1/アレル 1 12, アレル 1/アレル 2 22, アレル 2/アレル 2
43 表14 13 箇所の SNP と V/S 比との関連解析 SNP ID 近傍の 遺伝子名 男性 女性 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 遺伝子型による平均値 (kg/m2) リスクアレルに対する効果 11 12 22 β(標準誤差) P 値 11 12 22 β(標準誤差) P 値 rs1514175 TNNI3K 0.87 ± 0.4 0.84 ± 0.42 0.76 ± 0.27 0.018 ( 0.015) 0.24 0.45 ± 0.26 0.44 ± 0.26 0.39 ± 0.17 0.016 ( 0.015) 0.30 rs1555543 PTBP2 0.86 ± 0.46 0.84 ± 0.34 0.86 ± 0.42 -0.003 ( 0.015) 0.85 0.46 ± 0.15 0.45 ± 0.23 0.45 ± 0.27 -0.003 ( 0.016) 0.85 rs713586 ADCY3 0.90 ± 0.45 0.83 ± 0.37 0.88 ± 0.42 0.01 ( 0.012) 0.40 0.46 ± 0.25 0.43 ± 0.24 0.48 ± 0.28 -0.009 ( 0.011) 0.39 rs2943650 IRS1 0.65 ± 0.32 0.88 ± 0.45 0.85 ± 0.39 0.007 ( 0.018) 0.67 0.43 ± 0.19 0.48 ± 0.28 0.45 ± 0.25 -0.021 ( 0.019) 0.26 rs2112347 POC5 0.86 ± 0.36 0.84 ± 0.41 0.90 ± 0.45 0.002 ( 0.011) 0.84 0.42 ± 0.24 0.47 ± 0.27 0.45 ± 0.23 0.019 ( 0.011) 0.079 rs206936 NUDT3 0.84 ± 0.41 0.84 ± 0.41 0.87 ± 0.40 0.012 ( 0.011) 0.29 0.47 ± 0.26 0.43 ± 0.25 0.46 ± 0.26 -0.0002 ( 0.011) 0.99 rs10968576 LINGO2 0.84 ± 0.41 0.88 ± 0.40 0.9 ± 0.37 0.017 ( 0.014) 0.23 0.46 ± 0.27 0.44 ± 0.23 0.41 ± 0.21 -0.009 ( 0.014) 0.53 rs4929949 STK33 0.87 ± 0.37 0.86 ± 0.42 0.83 ± 0.40 0.008 ( 0.012) 0.48 0.48 ± 0.28 0.44 ± 0.25 0.44 ± 0.26 0.01 ( 0.011) 0.38 rs4771122 MTIF3 0.86 ± 0.41 0.85 ± 0.40 0.73 ± 0.40 -0.023 ( 0.013) 0.084 0.45 ± 0.27 0.45 ± 0.24 0.42 ± 0.22 -0.006 ( 0.013) 0.67 rs534870 SPRY2 0.84 ± 0.39 0.87 ± 0.43 0.83 ± 0.35 0.001 ( 0.011) 0.93 0.47 ± 0.26 0.45 ± 0.26 0.42 ± 0.23 0.017 ( 0.011) 0.14 rs2241423 MAP2K5 0.85 ± 0.38 0.88 ± 0.43 0.78 ± 0.37 -0.011 ( 0.012) 0.34 0.43 ± 0.25 0.47 ± 0.26 0.43 ± 0.25 0.021 ( 0.011) 0.073 rs2287019 QPCTL 0.84 ± 0.39 0.88 ± 0.44 0.79 ± 0.33 -0.004 ( 0.014) 0.80 0.44 ± 0.24 0.47 ± 0.28 0.49 ± 0.28 -0.014 ( 0.014) 0.32 rs3810291 ZC3H4 0.81 ± 0.35 0.86 ± 0.39 0.85 ± 0.42 -0.001 ( 0.012) 0.92 0.45 ± 0.22 0.43 ± 0.28 0.46 ± 0.24 -0.02 ( 0.013) 0.12 V/S 比は平均値±標準偏差で示し、P 値は年齢で補正した値を示した。 11, アレル 1/アレル 1 12, アレル 1/アレル 2 22, アレル 2/アレル 2
44 表15 13 箇所の SNP と血清パラメーターとの関連解析 SNP ID 遺伝子名 近傍の 血糖値 (mg/dL) インスリン (μU/mL) インスリン抵抗性指数 総コレステロール (mg/dL) β (標準誤差) P 値 (標準誤差) β P 値 (標準誤差) β P 値 (標準誤差) β P 値 rs1514175 TNNI3K ( 0.005) 0.000 0.95 ( 0.016) 0.017 0.29 ( 0.018) 0.013 0.49 ( 1.944) -0.118 0.95 rs1555543 PTBP2 ( 0.005) -0.002 0.67 ( 0.016) 0.027 0.095 ( 0.018) 0.024 0.20 ( 2.004) 0.193 0.92 rs713586 ADCY3 ( 0.004) -0.008 0.032 ( 0.012) 0.003 0.77 ( 0.013) -0.004 0.78 ( 1.419) 0.223 0.87 rs2943650 IRS1 ( 0.006) 0.006 0.33 ( 0.019) -0.024 0.20 ( 0.022) -0.02 0.34 ( 2.328) 1.879 0.42 rs2112347 POC5 ( 0.004) 0.001 0.75 ( 0.012) 0.003 0.8 ( 0.013) 0.003 0.84 ( 1.419) -3.16 0.026 rs206936 NUDT3 ( 0.004) 0.003 0.40 ( 0.012) -0.011 0.33 ( 0.013) -0.009 0.50 ( 1.427) -1.147 0.42 rs10968576 LINGO2 ( 0.005) 0.002 0.62 ( 0.015) -0.026 0.072 ( 0.017) -0.027 0.10 ( 1.779) 5.881 0.00097 rs4929949 STK33 ( 0.004) 0.004 0.31 ( 0.012) 0.003 0.77 ( 0.014) 0.006 0.63 ( 1.451) 1.056 0.47 rs4771122 MTIF3 ( 0.005) -0.009 0.053 ( 0.014) -0.024 0.083 ( 0.016) -0.035 0.028 ( 1.700) -1.415 0.41 rs534870 SPRY2 ( 0.004) 0.002 0.58 ( 0.012) 0.002 0.88 ( 0.014) 0.005 0.73 ( 1.468) 0.336 0.82 rs2241423 MAP2K5 ( 0.004) 0.005 0.22 ( 0.012) 0.004 0.73 ( 0.014) 0.008 0.57 ( 1.479) 1.492 0.31 rs2287019 QPCTL ( 0.005) 0.001 0.81 ( 0.015) 0.002 0.90 ( 0.017) 0.001 0.94 ( 1.806) 2.855 0.11 rs3810291 ZC3H4 ( 0.004) 0.000 0.95 ( 0.013) 0.000 0.98 ( 0.015) -0.001 0.93 ( 1.616) 0.537 0.74 データは平均値±標準偏差で示し、P 値は年齢、性別、BMI で補正した値を示した。
45 SNP ID 遺伝子名 近傍の 中性脂肪 (mg/dL) β HDL-コレステロール (mg/dL) 収縮期血圧 (mmHg) 拡張期血圧 (mmHg) (標準誤差) P 値 (標準誤差) β P 値 (標準誤差) β P 値 (標準誤差) β P 値 rs1514175 TNNI3K ( 0.012) -0.003 0.80 ( 0.753) 0.723 0.34 ( 0.905) -0.343 0.70 ( 0.629) -0.209 0.74 rs1555543 PTBP2 ( 0.012) -0.007 0.54 ( 0.774) 0.015 0.98 ( 0.943) -0.590 0.53 ( 0.653) -0.488 0.45 rs713586 ADCY3 ( 0.009) 0.008 0.33 ( 0.550) -0.769 0.16 ( 0.661) -1.243 0.060 -0.547 ( 0.46) 0.23 rs2943650 IRS1 ( 0.014) -0.02 0.16 ( 0.901) 1.036 0.25 ( 1.085) -0.205 0.85 ( 0.755) -0.180 0.81 rs2112347 POC5 ( 0.009) -0.005 0.55 ( 0.548) -0.859 0.12 ( 0.667) -0.107 0.87 ( 0.463) -0.373 0.42 rs206936 NUDT3 ( 0.009) 0.006 0.49 ( 0.554) -0.272 0.62 ( 0.665) 0.694 0.30 ( 0.462) 0.265 0.57 rs10968576 LINGO2 ( 0.011) 0.021 0.056 -0.385 ( 0.69) 0.58 ( 0.823) 0.835 0.31 ( 0.573) 0.154 0.79 rs4929949 STK33 ( 0.009) 0.020 0.021 ( 0.562) -0.648 0.25 ( 0.673) -0.235 0.73 ( 0.468) -0.126 0.79 rs4771122 MTIF3 ( 0.010) -0.005 0.65 ( 0.661) -0.179 0.79 ( 0.785) 0.426 0.59 ( 0.546) 0.313 0.57 rs534870 SPRY2 ( 0.009) 0.023 0.0094 ( 0.567) -1.865 0.0010 ( 0.68) 0.628 0.36 ( 0.473) 0.351 0.46 rs2241423 MAP2K5 ( 0.009) 0.001 0.95 ( 0.572) -0.050 0.93 ( 0.693) -0.410 0.56 ( 0.482) -0.146 0.76 rs2287019 QPCTL ( 0.011) -0.006 0.58 ( 0.702) -0.105 0.88 ( 0.849) 0.142 0.87 ( 0.590) 0.074 0.90 rs3810291 ZC3H4 ( 0.010) 0.013 0.17 ( 0.628) -0.028 0.96 ( 0.751) 0.781 0.30 ( 0.517) 0.626 0.23 rs1514175 TNNI3K ( 0.012) -0.003 0.80 ( 0.753) 0.723 0.34 ( 0.905) -0.343 0.70 ( 0.629) -0.209 0.74 データは平均値±標準偏差で示し、P 値は年齢、性別、BMI で補正した場合の値を示した。
46 図 15 NUDT3 と Akt シグナリングパスウェイ
47
第
5 章 血中アディポネクチン量に関連した SNP の検討
脂肪組織は外界の温度変化から断熱して体温を保持する機能や脂肪酸をエネ ルギーとして貯蓄すること、またホルモンを産生する機能も持つなど、恒常性 の維持に重要な組織である。食事によって摂取された糖質(ブドウ糖)は代謝 されエネルギーに変換されるが、高脂肪食や過食・運動不足により余ったエネ ルギーは、皮下脂肪、内臓脂肪の順番で蓄積される。内臓脂肪が過剰に蓄積す ると余剰エネルギーは、肝臓や筋肉に蓄積し異所性脂肪沈着を引き起こし、イ ンスリン抵抗性を示す。インスリン抵抗性の発症においては、脂肪細胞から分 泌されるアディポネクチンの役割も重要である。肥満者の脂肪細胞ではアディ ポネクチンの分泌が低下し、インスリン抵抗性と2 型糖尿病を引き起こす[4、5、 36、37]。アディポネクチンの分泌量低下は、異所性脂肪蓄積や高血糖や高血圧、 高脂血症などの様々な異常を導き、メタボリックシンドロームへと至ることが 知られている[3]。 近年のGWAS により、アディポネクチンレベルが内臓脂肪蓄積に加えて遺伝 素因によっても影響を受けることが報告されている。アディポネクチンレベル に影響する最も重要な遺伝素因は、アディポネクチンを産生するADIPOQ 遺伝 子そのものの多型であり、またADIPOQ 遺伝子の多型はアディポネクチンレベ ルだけではなく、インシュリン抵抗性や 2 型糖尿病とも関連していることが報 告されている[38-46]。多くの研究により、内臓脂肪面積も遺伝素因の影響を受 けることが報告されている[15-18、22、88]。 これらのことから、ADIPOQ 遺伝子は内臓脂肪蓄積と関連する有望な候補遺 伝子と考えられている[47、48]。しかし、ADIPOQ 遺伝子の多型と内臓脂肪面 積との関連を解析した研究はほとんどなく、また日本人集団を対象とした解析48 は行われていない。さらに、ADIPOQ 遺伝子の多型とインスリン抵抗性との関 連も決定的には立証されていない。 そこで本研究では、最近の欧米人、フィリピン人、韓国人、豪州人を対象と したGWAS 研究により見出された ADIPOQ 遺伝子の 7 箇所の SNP について、 日本人集団を用いて内臓脂肪面積やインスリン抵抗性との関連を調べた。
49
第
1 節 サンプルと対象 SNP の遺伝子型決定
サンプルは、2002 年から 2011 年に外来を訪れて定期健康診断を受けて、肥 満、高血圧、脂質異常症、2 型糖尿病などの治療を行った日本人集団から、1st セットとしてVFA、SFA のデータのある 1595 人(男性 731 人、女性 864 人)、 2nd セットとして VFA、SFA のデータのない 893 人(男性 383 人、女性 510 人)の、合計2488 人(男性 1114 人、女性 1374 人)を用いた(表 16)。体重に 影響を及ぼすことが予想される疾患(癌、腎臓病、循環器疾患、肝不全)や薬 物治療(インスリン、副腎皮質ステロイド、化学療法)を受けた者は除外した。 全ての対象者から書面によるインフォームドコンセント得て、実施計画は、京 都大学の倫理委員会により承認された(倫理委員会承認番号:G374)。 SNP は、欧米人、フィリピン人、韓国人、豪州人を対象とした GWAS によっ て見出さ れた ADIPOQ 遺伝子に存在する 7 箇所の SNP (rs6810075、 rs10937273、rs1648707、rs864265、rs182052、rs17366568、rs6773957)を対象とした。2488 人の血液から抽出したゲノム DNA(Genomix kit, Talent Srl,
Trieste,Italy)を鋳型として、第 4 章と同様に Multi-plex PCR 法とインベーダ
50 表16 対象者の臨床情報 1st セット 2nd セット 男性 女性 男性 女性 サンプル数 731 864 383 510 年齢 (歳) 48.2 ± 12.6 (39.0, 48.0, 59.0) 52.8 ± 11.4 (46.0, 54.0, 61.0) 60 ± 13.8 (52.0, 61.0, 71.0) 61.4 ± 11.7 (54.0, 62.0, 70.0) 体格指数 BMI (kg/m2) 29.7 ± 5.8 (26.3, 29.0, 32.0) 28.1 ± 5.3 (24.8, 27.1, 30.7) 26.2 ± 4.7 (23.0, 25.5, 28.4) 25.5 ± 4.1 (22.7, 25.2, 27.9) 内臓脂肪面積 VFA (cm2) 152.5 ± 66.3 (107.6, 142.7, 191.0) 104.7 ± 54.4 (65.4, 97.5, 136.2) 皮下脂肪面積 SFA (cm2) 204.9 ± 106.8 (136.5, 181.6, 245.5) 242.3 ± 98.8 (178.7, 226.3, 293.4) 血糖値 (mg/dL) 110.6 ± 33.5 (92.0, 100.0, 115.0) 109.1 ± 35.6 (90.0, 97.0, 112.8) 105.6 ± 25.7 (92.0, 99.0, 111.5) 99.9 ± 19.6 (89.0, 95.0, 104.8) インスリン (μU/mL) 13.7 ± 16.9 (6.3, 9.8, 15.7) 10.8 ± 10.3 (5.0, 8.1, 13.3) 12.7 ± 18.4 (3.9, 6.6, 13.7) 10.6 ± 11.7 (4.6, 6.7, 11.6) インスリン抵抗性指数 4.0 ± 7.1 (1.5, 2.5, 4.2) 3.1 ± 3.8 (1.2, 2.1, 3.5) 3.7 ± 7.1 (0.9, 1.7, 3.4) 2.8 ± 3.5 (1.1, 1.6, 2.9) 血中アディポネクチン (g/mL) 6.1 ± 3.1 (4.2, 5.5, 6.9)* 9.8 ± 5.0 (6.4, 8.8, 11.7)§ 肥満 608 (83.2%) 624 (72.2%) 217 (56.7%) 268 (52.5%) 耐糖能異常症 243 (33.2%) 258 (29.9%) 107 (27.9%) 92 (18.0%) 2 型糖尿病 152 (20.8%) 174 (20.1%) 88 (23.0%) 75 (14.7%) 脂質異常症 403 (55.1%) 319 (36.9%) 157 (41.0%) 143 (28.0%) 高血圧症 404 (55.3%) 490 (56.7%) 244 (63.7%) 288 (56.5%) 2 型糖尿病治療 98 (13.4 %) 106 (12.3%) 27 (7.0 %) 24 (4.7 %) データは平均値±標準偏差(第 1 四分位点、中央値、第 3 四分位点). 血中アディポネクチンについては、男性(*)412 人、女性(§)533 人
51
第
2 節 脂肪分布パラメーターと血清パラメーターの関連解析
1595 人の VFA と SFA は CT [26]による測定値を用いた。V/S 比は VFA/ SFA として解析した。 脂肪分布パラメーター(BMI、VFA、SFA、V/S 比)と血清パラメーター(血 糖値、インスリン、インスリン抵抗性指数、血中アディポネクチン量)を従属 変数として、遺伝子型(リスクアレル保有数により0、1、2 と変換)、性別、年 齢、BMI を説明変数として多重線形回帰分析を行った。インスリン抵抗性はイ ンスリン抵抗性指数(HOMA-IR)で評価した。
第
1 項 アディポネクチン
アディポネクチンは、1996 年に大阪大学分子制御内科学の松澤らのグループにより脂肪組織中に特異的に発現する遺伝子 apM1(adipose most abundant
gene transcript)として同定されたサイトカイン(アディポサイトカイン)で あり、244 個のアミノ酸よりなる分泌タンパク質である[86、87]。 近年、アディポネクチンは様々な生理活性を有することが報告されている。 マウスにアディポネクチンを投与することによって、糖尿病が改善することが 見出された[50]。また、アディポネクチン低下と肥満や糖尿病、動脈硬化との関 連が報告されている[5、37、49、51]。 ヒト血清・血漿や脂肪細胞抽出液または培養上清中のアディポネクチンは、 酵素免疫測定法 (Enzyme-Linked Immunosorbent Assay: ELISA)を用いて 測定することができる。
52
第
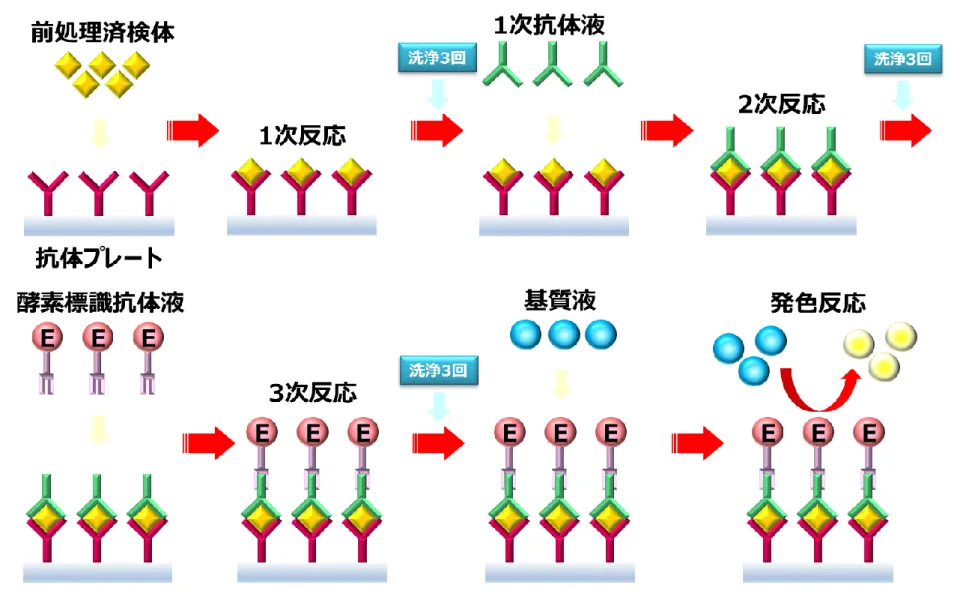
2 項 ヒトアディポネクチン ELISA キット(大塚製薬株式会社)
① 原理と測定 抗ヒトアディポネクチンモノクローナル抗体固相プレート(抗体プレート) に、希釈済みサンプルを加えて反応させると、アディポネクチンが抗体プレー トに結合する(第1 反応)。第 1 抗体液(抗ヒトアディポネクチン抗血清(ウサ ギ))を反応させ(第 2 反応)、酵素標識抗体液(西洋ワサビペルオキシダーゼ 標識抗ウサギIgG ポリクローナル抗体(ヤギ))を反応させる(第 3 反応)。基 質液を加えて発色させ(発色反応)、Infinite 200 PRO(テカンジャパン株式会 社)を用いて、吸光度 450 nm で測定し、同時に測定した標準液の吸光度から 検体中のアディポネクチン濃度を算出した(図16)。 ② 検体の前処理 血漿サンプル10 μL を検体前処理液 90 μL と混和後、ヒートブロック等を用 いて100℃で 5 分間加熱した(10 倍希釈)。さらに検体希釈液を 900 μL 加え混 和した(100 倍希釈)。100 倍希釈した血漿サンプル 20 μL を検体希釈液 1 mL と混和し、前処理済みの血漿サンプルとした(5,100 倍希釈)。 ③ 試薬の調整 (1) 洗浄液 洗浄用原液全量(40 mL)に精製水を 960 mL の割合で混和し調整した。 洗浄用原液に結晶が析出している場合は、加温して溶解後に調整し、調整後 は4℃で保存した。53 (2) 検体希釈液 検体希釈用原液全量(50 mL)に精製水を 200 mL の割合で混和し調整した。 調整後は4℃で保存した。 (3) 標準液 12.0 ng/mL は標準品リコンビナントヒトアディポネクチン 12.0 ng/mL を使 用し、他の標準液は標準品リコンビナントヒトアディポネクチン12.0 ng/mL を 検体希釈液で段階希釈して、作成した。0 ng/mL は検体希釈液を使用した。検 量線は12.0 ng/mL、6.0 ng/mL、3.0 ng/mL、1.5 ng/mL、0.75 ng/mL、0.375 ng/mL、0 ng/mL とした。 (4) 酵素標識抗体液 酵素標識抗体希釈液12 mL に酵素標識抗体原液を 60 μL の割合で混和し調整 した。第2 反応の直前に調整し、速やかに使用した。 (5) 基質液 基質液B 6 mL に基質液 A 6 mL の割合で混和し調整した。発色反応の直前に 調整し、速やかに使用した。 (6) 検体前処理液 析出していた場合は、泡立てないように加温して溶解後に使用した。 ④ 測定方法 (1) 各構成試薬を室温に戻した。 (2) 洗浄液、検体希釈液、各濃度の標準液を調整した。
54 (3) 抗体プレートのアルミラミネート袋を開封した。 (4) 洗浄液を抗体プレートの各ウェルに約 350 μL ずつ加えた後、ウェルの液を 完全に除去した。抗体プレートを逆さにしてキムタオルに軽く叩き付け、ウ ェル内に残った洗浄液を除去した。 (5) 各濃度の標準液及び前処理済みの検体を各ウェルに 100 μL ずつ加えた。標 準液は各測定時に、各抗体プレートについて必ず測定した。 (6) プレートシールで抗体プレートをカバーし、室温で 60 分間静置反応させた。 (7) 液はねしないように、注意して抗体プレートからプレートシールを取り除き、 ウェルの液を完全に除去した。洗浄液を抗体プレートの各ウェルに約 350 μL ずつ加え、再度速やかに除去した。この操作を 2 回繰り返した後、抗体 プレートを逆さにしてキムタオルに軽く叩き付け、ウェル内に残った洗浄液 を除去した(洗浄3 回)。 (8) 第 1 抗体液を抗体プレートの各ウェルに 100 μL ずつ加えた。 (9) プレートシールで抗体プレートをカバーし、室温で 60 分間静置反応させた。 (10) 液はねしないように、注意して抗体プレートからプレートシールを取り 除き、ウェルの液を完全に除去した。洗浄液を抗体プレートの各ウェルに約 350 μL ずつ加え、再度速やかに除去した。この操作を 2 回繰り返した後、 抗体プレートを逆さにしてキムタオルに軽く叩き付け、ウェル内に残った洗 浄液を除去した(洗浄3 回)。 (11) 酵素標識抗体液を抗体プレートの各ウェルに100 μL ずつ加えた。 (12) プレートシールで抗体プレートをカバーし、室温で 60 分間静置反応さ せた。 (13) 液はねしないように、注意して抗体プレートからプレートシールを取り 除き、ウェルの液を完全に除去した。洗浄液を抗体プレートの各ウェルに約
55 350 μL ずつ加え、再度速やかに除去した。この操作を 2 回繰り返した後、 抗体プレートを逆さにしてキムタオルに軽く叩き付け、ウェル内に残った洗 浄液を除去した(洗浄3 回)。 (14) 基質液を抗体プレートの各ウェルに100 μL ずつ加えた。 (15) 室温で 15 分間静置反応させた後、反応停止液を抗体プレートの各ウェ ルに100 μL ずつ加えた。 (16) Infinite 200 PRO(テカンジャパン株式会社)を用いて、吸光度 450 nm で測定し、同時に測定した標準液の吸光度から検体中のアディポネクチン濃 度を算出した。 製品番号:410614 製品名 :ヒトアディポネクチンELISA キット(大塚製薬株式会社)
56 図 16 ヒトアディポネクチン ELISA キットの測定原理と方法
57
第
3 項 統計解析
ハーディーワインバーグ平衡は2テストで評価した[28]。多重線形回帰分析は R software (https://www.r-project.org/)を使用し、ボンフェローニの補正を 使用してP 値は 0.0071(0.05/7 箇所の SNP)未満の値を統計学的に有意である とした。1st セット、2nd セットそれぞれの解析と、1st セットと 2nd セットの メタ解析を行った。また、脂肪分布は性的特異性があることが知られているた め[29]、男女別に解析を行った。 日本人はインスリンレベルが低いことが知られているので[52]、対象者の BMI については、広いレンジで関連解析を行った。第
4 項 ハプロタイプ解析
ADIPOQ 遺伝子のハプロタイプと HOMA-IR との関連を調べるために、 Haplo.Stats (http://mayoresearch.mayo.edu/mayo/research/schaid_lab/software.cfm)を 使用して解析を行った。58
![図 1 内臓脂肪蓄積と異所性脂肪沈着 [1]](https://thumb-ap.123doks.com/thumbv2/123deta/7038693.1201668/5.1262.185.1086.125.803/図1内臓脂肪蓄積と異所性脂肪沈着1.webp)
![図 3 SNP を活用した集団構造の解析 [90]](https://thumb-ap.123doks.com/thumbv2/123deta/7038693.1201668/8.1262.752.1068.129.655/図3SNPを活用した集団構造の解析9.webp)