内モンゴル塩湖から単離されたHalorubrum sp.
ejinoorの持つロドプシン類タンパク質の研究
著者
潮 洛蒙
学位名
博士(工学)
学位の種別
課程博士
報告番号
甲第386号
研究科・専攻
創成機能工学専攻
学位授与年月日
2016-03-23
URL
http://hdl.handle.net/10258/00008931
博士学位論文
内モンゴル塩湖から単離された
Halorubrum
sp. ejinoor の持つロドプシン類
タンパク質の研究
2016 年 3 月 22 日
室蘭工業大学大学院 工学研究科
創成機能工学専攻 博士後期課程
潮洛蒙
2
要旨
細菌ロドプシン類はレチナールを発色団として持ち、光を受容すると、まず 発色団が構造変化を起こし、次いでタンパク質の構造変化を誘発する。細菌ロ ドプシンは世界各地から見つかっている。中国内モンゴル自治区には多くの塩 湖が存在するが、それらの塩湖から細菌ロドプシン類が検出されたという報告 はなかった。私は中国の内モンゴルにある塩湖(エジノル)から持つ細菌ロド プシンタンパク質遺伝子を持つ好塩菌を単離して調べた。 中国内モンゴル・エジノル塩湖の湖水をフィルター濾過して細菌を集め、好 塩菌を寒天培地に広げ、そのコロニーを分離した。単離されたコロニーからゲノムDNAを抽出し、16S rRNA遺伝子を増幅した。増幅された16S rRNA遺伝子配列
を決定したところ、その配列はHalorubrum chaoviator のものと最も相同性が
高かったので、単離した好塩菌をHalorubrum sp. ejinoor (He)と名付けた。He
からゲノムDNAを抽出し、全ゲノム解析を行った。その結果、三種類の細菌ロド
プシン類タンパク質遺伝子、BR-like(HeAR)、HR-like(HeHR)、SRII-like(HeSRII)
を見つけた。それらのタンパク質遺伝子に対してQuantitative RT-PCRを行った
結果、菌体中でのmRNAの比率がHeAR : HeHR : HeSRII : HeHtrII = 30 : 10 : 1 :
1であることを明らかにした。次に、菌体膜から凍結融解法により菌体膜小胞を 作製し、その光駆動ポンプ活性を調べた。菌体膜小胞を4M NaClに懸濁し、590nm の光で照射すると、外液のアルカリ化が観察された。菌体膜小胞液に10 uM CCCP を入れると光照射に伴う外液のアルカリ化は大きくなった。10 uM TPP存在下で は外液の酸性化が観察された。菌体膜小胞をクロライドイオンがない条件(3M NaNO3)で光照射すると外液の酸性化が観察された。以上の結果より菌体膜小胞 にはHRと同様のクロライドイオンポンプ活性とBRと同様のプロトンポンプ活性 があると結論できた。Halobacterium salinarum(Hs)の紫膜を分離するのと同
3 様に60~30%のショ糖溶液のステップ密度勾配で超遠心を行い、菌体膜の分離 を試みた。Heの膜はHsのように明瞭に2層には分かれなかった。このことより、 HeARは紫膜様の膜構造はとらないものと結論された。レーザー閃光分光法で菌 体膜の光反応を410 nm、670 nm、540 nm、570 nmの波長で追跡したところ、BR のようなM中間体の生成・崩壊を示す吸収変化を観察できた。さらにクロライド イオンがある条件下と無い条件下での510nmでの吸光度変化が大きく異なるこ とからHRの存在が示唆された。次に発現精製したタンパク質を得て、より詳細 にそれらの性質を調べることにした。
HeAR を大腸菌で発現した。Ni カラムより精製した AR は紫色で、SDS-PAGE 解
析では分子量が 27 kDa であり、予測した分子量と一致した。吸収極大波長は 555 nm であった。CD スペクトルより精製した AR が 0.15% DDM を含むバッファ中で 三量体構造を持つことが分かった。精製した AR は明暗順応を行うので、発色団 レチナールの異性体構造を調べた。また、レーザー閃光分解解析から光反応を 追跡したところ、(K)、M、N、O 中間体を持ち、光反応サイクル時間が 70 ms で あることを明らかにした。 HeAR 遺伝子を発現させた大腸菌では、光照射に伴う、 外液の酸性化が観察された。この結果は、HeAR がプロトンポンプ細菌ロドプシ ンであることを示している。
4
PhD Thesis
Study on the microbial rhodopsins of
Halorubrum
sp. ejinoor isolated from Ejinoor salt lake in
Inner Mongolia of China
Chaoluomeng
5
Abstract
Microbial rhodopsins belong to the large family of retinal protein, a
family of photoactive protein which has seven trans-membrane helices and
a retinal molecule binding to a lysine residue in the seventh helix via
the Schiff base as the photoactive center. Because of structural simplicity
and functional diversity, microbial rhodopsins have been an excellent model
system for structural biology.
In this study, a bacterial strain was collected and identified from a
salt lake in Inner Mongolia of China. According to the sequence of 16S rRNA
gene of the bacterium the strain belongs to Halorubrum genus and was named
Halorubrum sp. ejinoor (He). Three kinds of microbial rhodopsin genes,
BR-like (HeAR), HR-like (HeHR) and SRII-like (HeSRII) were identified in
He by a whole genome DNA analysis of the bacterium. The mRNAs of three types
of microbial rhodopsin genes were detected by RT-PCR and their amounts were
determined by quantitative RT-PCR. The amount of mRNA of these genes in
He was HeAR : HeHR : HeSRII = 30 : 10 : 1. Measurements of light-induced
ion transportation of membrane vesicles suggest the existence of a proton
pumping activity and a chloride pumping activity in membrane vesicles of
He. He membrane fraction was isolated from cell membrane of the bacterium
by sucrose density gradient centrifugation method similar to that for
isolation of purple membrane from Halobacterium salinarum. The kinetics
of the light-induced absorbance changes of the membrane fraction of He
suggested the presence of M-like intermediate (HeAR) and L-like
6
HeAR, a light-driven proton pump of Halorubrum sp. ejinoor was
heterologously expressed in Escherichia coli. The expressed HeAR showed
purple colour in the E. coli membrane and was purified by Ni-chelate
chromatography. The purified HeAR shows a light-dark adaptation. After
illumination, the purified HeAR showed a spectral redshift from 555 to 565
nm, suggesting an increase in the all-trans retinal content. The HPLC
analysis revealed that the spectral shift accompanies with the change in
retinal contents from 66% all-trans and 34% 13-cis in the dark to 85%
all-trans and 15% 13-cis after illumination. The time courses of the
absorbance change of HeAR suggested the presence of (K), M, N and O
intermediates similar to those of BR-system. The half-life of the M
intermediate was estimated as 0.001 s, faster than that in BR (0.005 s).
But the photocycling rate was slower than BR. The measurements of
light-induced pH changes of the E. coli cell suspensions containing HeAR
7
略語リスト
BR: Bacteriorhodopsin from Halobacterium salinarum
HR: Halorhodopsin from Halobacterium salinarum
SRI: Sensory rhodopsin I from Halobacterium salinarum
SRII: Sensory rhodopsin II from Halobacterium salinarum
AR: Archaerhodopsin
ChR: Channelrhodopsin
He: Halorubrum sp. ejinoor
Hs: Halobacterium salinarum
NpHR: Halorhodopsin from Natronomonas pharaonis
NpSRII: Sensory rhodopsin II from Natronomonas pharaonis
HeAR: Archaerhodopsin from Halorubrum sp. ejinoor
HeHR: Halorhodopsin from Halorubrum sp. ejinoor
HeSRII: Sensory rhodopsin II from Halorubrum sp. ejinoor
HeHtrII: Transducer protein from Halorubrum sp. ejinoor
HsPM: Purple membrane of Halobacterium salinarum
HsRM: Red membrane of Halobacterium salinarum
HeBM: Brown membrane of Halorubrum sp. ejinoor
CCCP:carbonyl cyanide m-chlorophenylhydrazone
TPP+:tetraphenylphosphonium
PCR: Polymerase Chain Reaction
RNA: Ribonucleic Acid
mRNA: messenger RNA
8 cDNA: complementary deoxyribonucleic acid
RT-PCR: Reverse Transcription PCR
Quantitative RT-PCR: Quantitative Real Time-PCR
NCBI:National Center for Biotechnology Information
EDTA: ethylenediaminetetraacetic acid
E. coli: Escherichia coli
IPTG: isopropyl-1-thio-β-galactoside
SDS-PAGE: sodium dodecyl sulfate-polycrylamid gel electrophoresis
CD: Circular Dichroism
DDM: n-dodecyl-β-D-maltoside
9
目次
第一章 序論... 12
1.1 マイクロバイアルロドプシン ... 12 1.1.1 古細菌ロドプシン ... 12 1.1.2 光駆動ポンプと光反応... 18 1.2 オプトジェネテクスへの応用 ... 21 1.3 本研究の目的 ... 22第二章
Halorubrum
sp.ejinoor のロドプシン類タンパク質 ... 23
2.1 好塩菌の単離 ... 24 2.1.1 方法 ... 25 2.1.1.1 試料 ... 25 2.1.1.2 菌の培養 ... 25 2.1.2 結果 ... 26 2.2 16S rRNA 遺伝子配列による菌の同定 ... 27 2.2.1 方法 ... 27 2.2.1.1 ゲノム DNA 抽出 ... 27 2.2.1.2 PCR ... 28 2.2.1.3 目的遺伝子配列をベクターへサブコロニング ... 30 2.2.1.4 プラスミド DNA 抽出 ... 31 2.2.1.5 DNA 配列決定 ... 31 2.2.2 結果 ... 32 2.3 Halorubrum sp. ejinoor からのロドプシン類タンパク質の探索 ... 35 2.3.1 方法 ... 35 2.3.1.1 PCR ... 35 2.3.1.2 ゲノム DNA 解析 ... 35 2.3.2 結果 ... 362.4 RT-PCR 解析と Quantitative Real Time-PCR 遺伝子発現解析 ... 41
10
2.4.1.1 RNA 抽出 ... 41
2.4.1.2 RT-PCR と Quantitative Real Time-PCR ... 41
2.4.2 結果 ... 43 2.5 菌体膜小胞の光駆動イオンポンプ活性... 45 2.5.1 方法 ... 45 2.5.1.1 菌体膜小胞作製 ... 45 2.5.1.2 光照射に伴うプロトン濃度変化の測定 ... 45 2.5.2 結果 ... 46 2.6 菌体膜の光反応 ... 49 2.6.1 方法 ... 49 2.6.1.1 菌体膜の分離 ... 49 2.6.1.2 レーザー誘起吸光度変化の比較 ... 49 2.6.2 結果 ... 50 2.7 まとめ ... 54
第三章
Halorubrum
sp.ejinoor の持つアーキロドプシンの大腸菌
による発現精製 ... 55
3.1 アーキロドプシンの発現精製 ... 56 3.1.1 方法 ... 56 3.1.1.1 発現ベクターの作製 ... 56 3.1.1.2 アーキロドプシン遺伝子配列の発現ベクターへの導入 ... 61 3.1.1.3 大腸菌によるアーキロドプシンの発現 ... 61 3.1.1.4 発現アーキロドプシンの可溶化と Ni アフィニテイカラムによる精製 62 3.1.2 結果 ... 62 3.2 アーキロドプシンの吸収スペクトルと CD スペクトル... 64 3.2.1 方法 ... 64 3.2.2 結果 ... 64 3.3 アーキロドプシンの明暗順応と発色団の解析... 66 3.3.1 方法 ... 6611 3.1.1.4 明暗順応 ... 66 3.1.1.2 発色団レチナールの抽出および HPLC 解析 ... 66 3.3.2 結果 ... 67 3.4 アーキロドプシン光反応サイクルアーキロドプシンの光反応 ... 71 3.4.1 方法 ... 71 3.4.2 結果 ... 72 3.5 アーキロドプシンの光駆動プロトンポンプ機能 ... 76 3.5.1 方法 ... 76 3.5.2 結果 ... 76 3.6 まとめ ... 77
第四章 結論および考察 ... 78
参考文献 ... 82
謝辞 ... 91
研究業績 ... 93
12
第一章 序論
1.1 マイクロバイアルロドプシン
1.1.1 古細菌ロドプシン
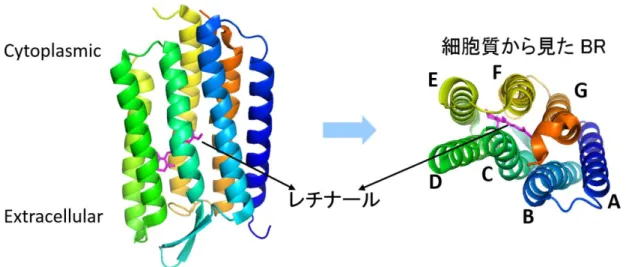
古細菌ロドプシン類は古細菌(Archaea)に属する高度好塩菌より単離された 7本の a-へリックスから構成され、発色団レチナールを持っている光受容膜タ ンパク質である。それらはレチナールに吸収された光をエネルギー源として、 様々な機能を持つ。古細菌ロドプシンの中で最初に発見されたのがバクテリオ ロドプシン(BR)である(図 1.1)[1]。BR は 1971 年 Dieter Oesterhelt 博士らが高度好塩菌Halobacterium halobium,
(現在は Halobacterium salinarum というの紫膜から発見した(図 1.2)。
図 1.1: BR の立体構造を表す。左図: X 線結晶構造解析から明らかにされたレチ ナールを含んだ BR の立体構造モデル。右図:細胞質側から見た BR の7本のαヘ リックス。
13 図 1.2 : Halobacterium salinarum の走光性と光受容ロドプシン類タンパク質の 主な機能を表す。A: センサリーロドプシンがバクテリアの示す走光性の光受容 体として働いている。菌体膜に紫色の膜(BR)と赤色の膜(主としてバクテリオ ルべリンの色)があり、BR が光を受容することでプロトン濃度勾配が発生、それ を利用して ATP を合成している。紫膜は菌体膜中では特異的な二次元構造をもち、 一方向に並んでいる。B: ショ糖密度勾配遠心より分離した菌体膜(赤膜と紫膜)。 C: BR の三量体の結合より形成されている紫膜。D: 三量体は三分子の BR と脂質 よりつくられている。E: 三量体分子間のレチナール相互作用に由来する CD スペ クトルと吸収スペクトル。
14 培養した好塩菌を低塩濃度溶液に移して細胞を破砕し、遠心分離より膜断片を 集め ショ糖密度勾配遠心にかけると、赤色の膜断片と紫色の膜断片が得られ る(図 1.2B)。赤色の膜断片が bacterioruberin と呼ばれるカロテノイドを含む 膜断片である。紫膜(Purple membrane)を電気泳動で調べたところ、ただ一種 類のタンパク質 BR を含んだ膜であることがわかった[1, 2]。BR は光駆動外向き プロトンポンプであり、プロトンの濃度勾配を生成することに伴う ATP を合成 してエネルギーとして高度好塩菌に利用される[3]。Halobacterium salinarum は BR を大量に合成しているため菌体が紫色をしており、酸素が低い条件で最も 大量の BR を合成することを分かっている。つまり、この菌は BR を利用して光 合成をすることで光エネルギーを利用できる。BR は光を受容する発色団レチナ ールをもつため可視光域に吸収極大波長を持っている。BR は明暗順応を行い、 それぞれ吸収極大波長が異なる(明順応型:568nm;暗順応型:558nm)。暗所で は、レチナールの異性体組成は all-trans : 13-cis = 1:1 である。光照射によ って 13-cis が all-trans に変換する。これを明順応と呼び、明順応型を暗所に 置くと、暗順応型に戻る[4]。 1971 年 BR に続き、Halobacterium salinarum から内向きクロライドイオンポ ンプであるハロロドプシン(HR; 1977 年)[5, 6]、正の走光性センサーである センサリーロドプシン I(SRI; 1982 年)、負の走光性センサーであるセンサリ
ーロドプシン II(SRII; 1985 年)が発現された[7]。Halobacterium salinarum
は4種類の異なる機能をもつロドプシン類をもつことが明らかとなった(図
1.3)。HR は内向き光駆動クロライドイオンポンプとして働く光受容膜タンパク
質である。HR も BR と同じように明暗順応を示し、暗順応型の HR はクロライド
イオンと結合しやすく、その結合に伴い、吸収極大は 10 nm の長波長シフトを
15 図 1.3 : Halobacterium salinarum 発現したマイクロバイアルロドプシン類 タンパク質とそれらの機能を表す。 の解離定数は、数 mM ~ 20mM 程度であることがわかった。つまり、暗順応型の HR は生理的環境である高塩濃度条件 下では、クロライドイオンと結合してい る[8, 9]。
SRI と SRII は光情報を膜結合型の伝達タンパク質(HtrI, HtrII)に受け渡す。
センサリーロドプシンは光忌避反応の光受容体である。菌体内のセンサリーロ
ドプシンが光を受容すると HtrI や HtrII を活性化し、続いて、キナーゼ(CheA)
の活性化、レスポンスレギュレター(CheY)のリン酸化により鞭毛回転方向を
制御している[10-12]。センサリーロドプシンは伝達タンパク質と結合していな
いとプロトンポンプとして機能することが知られている[13]。Halobacterium
salinarum 菌体膜でのこれらのタンパク質の存在量比は(BR : HR : SRI : SRII)
16 へリックスにあるリジン残基とレチナールがシッフ塩基結合を形成している。 光を受容するとレチナールの構造が変わり、イオンポンプや光センサーとして 働いている。 今まで、古細菌から始まって徐々に新規な細菌ロドプシン類が発見されてき た。大きなグループに分けると光駆動イオンポンプと光センサーになっている (図 1.4)。光駆動プロトンポンプ類ロドプシンのなかで良く研究されているのは BR とHalorubrum 属から発見されたアーキロドプシン(AR)類である[15-19]。 光駆動クロライドイオンポンプには HR と Natronomonas pharaonis 由来の
HR(NpHR)がある[20, 21]。SRII では、SRII と Natronomonas pharaonis から発
見された SRII がある[22]。それらのアミノ酸配列は相当異なるが、構造的には 非常に類似している。プロトンポンプ型では、レチナール結合ポケットにかか わるアミノ酸が 22 個あり(BR)、プロトンポンプ機能に重要といわれている。 BR のシッフ塩基からのプロトンアクセプターとして機能するアミノ酸 Asp85 は、 HR では、Thr111 (NpHR:Thr126) になっている。HR のシッフ塩基の対イオンと してクロライドイオンが結合している。BR の Asp85 を Thr に変換することでク ロライドイオンポンプに変換できる[23]。一方、最近の研究では、HR のいくつ かのアミノ酸を変換してもプロトンポンプにはならなかった[24]
17
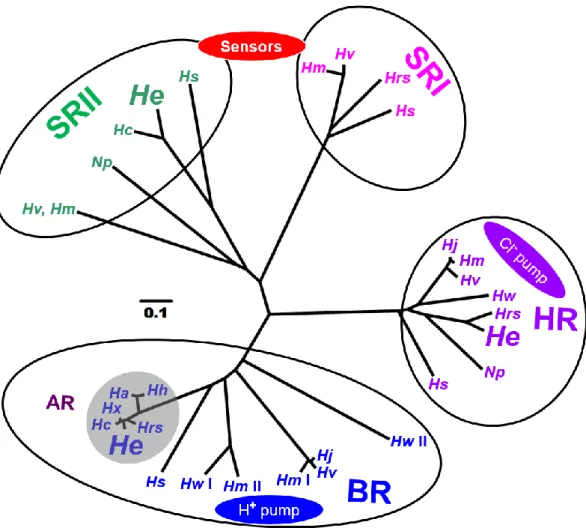
図 1.4 : マイクロバイアルロドプシン類タンパクの質分子系統樹とそれらの
Accession number (NCBI data base)。青:光駆動ポンプ;赤:光受容イオン チャネル;黄;光センサー
18
1.1.2 光駆動ポンプと光反応
最も研究されている BR では、光を受容するとレチナールが all-trans から 13-cis に異性化して、いくつかの中間体を経て元に戻る光化学反応サイクルを 行う。一回の光反応サイクルに応じて一個のプロトンを細胞膜内から外側へ輸 送する。光反応中間体はそれぞれの特徴的な吸収極大波長をもっている。光反 応とプロトンの輸送の関係を簡単に述べる。まず、BR が光を受容するとシッフ 塩基の pKa が下がり、シッフ塩基のプロトンがアクセプターになる脱プロトン 化しているアクセプター、Asp85 に渡される。この移動に伴ってプロトン放出複合体(Glu194, Glu204, Arg82 および水分子から構成されている)からプロトン
が細胞外側へ放出される。これが L から M への段階で起こる。次いで、プロト ン化しているドナー、Asp96 がプロトンを脱プロトン化しているシッフ塩基に渡 す(M から N)。この時、Asp85 の pKa が上がり、プロトンの逆流が起こらない ようにしている。そのあと、ドナー残基、Asp96 が細胞質からプロトンを受け取 り、レチナールが all-trans へ異性化する(N から O)。最後にプロトン化して いるアクセプターAsp85 がプロトンを放出複合体に渡し、もとの状態に戻る(O から BR)[8, 9]。 HR では、BR と同様の光反応に応じて、プロトンではなくクロライドイオンを細 胞外側から内側へ輸送する。BR のプロトンアクセプター、Asp85 は、HR では、 Thr111 になる。プロトンを受け取る残基がないので光反応に伴うシッフ塩基の 脱プロトン化が起こらない。NpHR の光反応では、K (598 nm), L1 (520 nm), L2 (520 nm), N (510 nm), O (605 nm)とNpHR` (575 nm)中間体がある。BR 光反応 サイクルに見られる短波長に吸収をもつ M 中間体がない。N 中間体から O 中間体 が生成する時にクロライドイオンが細胞内側へ放出され、O 中間体からNpHR`中 間体へ戻るときにクロライドイオンを細胞外側から取り込んでいる。HR では N
19 図 1.5 : BR と HR のイオンポンプ機能。左図:BR のプロトンポンプ機能過程 を表す。まず、Asp85 がシッフ塩基からプロトンをもらう(①)。次にプロ トン放出複合体よりプロトンを排出(②)。その後、Asp96 のプロトンをシ ッフ塩基に渡す(③)。細胞内からプロトンを Asp96 に渡す(④)。最後に Asp85 のプロトンを複合体に渡して(⑤)一サイクルが終わる。右図:HR の クロライドイオンポンプ過程。HR では BR の Asp85 に応じて Thr111 がクロラ イドイオンと結合している。光受容することでまず、Thr111 がクロライドイ オンをシッフ塩基に渡す(①)。その後、外のクロライドイオンを複合体が もらう(②)。シッフ塩基から細胞質側へトランスポートする(③)。最後 に複合体から Thr111 にクロライドイオンを渡して(④)光反応サイクルが終 わる。 と O 中間体がクロライドイオン濃度依存的に平衡関係にあると考えられている。 クロライドイオン増加に伴い、平衡が N へ移動し、逆にクロライドイオンを減 らすことで N 中間体が減っていくことが分かっている。N 中間体はクロライドイ オンと結合している状態、O 中間体は放出後の状態と言われている[9]。
20 図 1.6 : BR と HR の光反応。左図:BR が光を受容すると K, L, M, N, O 中間体を 経て元に戻っている。それらの中間体が特徴的な吸収極大波長を持つことを表 す。プロトンを L から M になるとき排出して、N から O になるときプロトンを受 け取っている。プロトンポンプ機能に重要な M 中間体を黄色で表した。右図: HR は BR と似た光反応を行い、元に戻る。クロライドイオンは N から O になるとき 離れ、O から HR`になるときクロライドイオンを受け取られる。クロライドポン プ機能に重要な N 中間体を赤で表した。
21
1.2 オプトジェネテクスへの応用
光を受容してイオンを駆動させるチャネルまたはイオンポンプタンパク質を コードする遺伝子を細胞に導入して、光照射により細胞の機能を制御および計 測する手法が最近開発され、オプトジェネテクスと名付けられた。 現在、オプトジェネテックスによく使われているロドプシン類には、光開閉 イオンチャネルとして働くチャネルロドプシン、光駆動クロライドイオンポン プである HR、光駆動プロトンポンプである BR と AR とがある。実験例としては、 H, K, Ca, Na イ オ ン を 透 過 さ せ る チ ャ ネ ル で あ る チ ャ ネ ル ロ ド プ シ ン (Channelrhodopsin; ChR)[25]を光刺激することで神経細胞を興奮させる[26]。 ChR と光ファイバを組み合わせることによって、生きているマウスの睡眠覚醒行 動を光によって制御することに成功した[27, 28]。今、ChR は、神経回路だけで はなく、疾患治療の基礎研究や応用にも使用されている[29, 30]。クロライド イオンポンプである NpHR とプロトンポンプである AR は神経細胞でよく発現さ れ、細胞毒性も低いため、神経興奮を制御する目的で使われている[31, 32]。 これらの研究に用いるために、光によって神経活動を脱分極と過分極の双方向 にコントロール可能な光受容タンパク質(イオンチャネルとプロトンポンプ) が開発されている[33, 34]。ChR が光受容することで陽イオンが細胞内に流入し、 神経細胞を脱分極させる[25, 35]。AR はプロトンを細胞外へ排出することで神 経細胞を過分極させる[32]。しかしながら、ChR と AR がどのようにイオンを透 過させ、AR が輸送するかについて詳しい分子機構は分かっていない。それらの ロドプシンの働くメカニズムを詳しく解明して、新機能を付与した変異タンパ ク質の作製、および新規なロドプシン類を発見することでオプトジェネテクス への応用にも貢献できると考えている。22
1.3 本研究の目的
マイクロバイアルロドプシン類は古細菌に属する高度好塩菌より最初に見出 された光受容タンパク質である。ロドプシン類の光化学的性質と特定のアミノ 酸の関係についての研究はよく進んでいるが、まだ分かっていないところも多 い。特にそれらのタンパク質の吸収極大の決定機構については古くからの課題 であるのに、未だ解明されていない。マイクロバイアルロドプシン類遺伝子を 持つ細菌は塩湖、海水など広い範囲から見つけられている。近年、中国のチベ ット、新疆の塩湖からマイクロバイアルロドプシン類を合成している菌をみつ けたという報告があった[16, 36]。しかし、中国内モンゴルの塩湖から見つけ たという報告は無かった。 本研究では、内モンゴルの塩湖の好塩菌がマイクロバイアルロドプシン類タ ンパク質遺伝子を持つかどうかを調べた。塩湖から新規なHalorubrum 属の菌を 単離して、Halorubrum sp. ejinoor と名付けた。その菌から三種類のマイクロ バイアルロドプシン類タンパク質遺伝子を発見した。そこで、1)Halorubrum sp. ejinoor 菌体膜を試料としてイオンポンプ活性や光反応を検討すること、2) それらのタンパク質を大腸菌で発現・精製し、精製したタンパク質を用いて、 それらの構造・機能を調べることを目的とした。23
第二章
Halorubrum
sp.ejinoor の ロ ド プ
シン類タンパク質
中国内モンゴルエジノル塩湖からマイクロバイアルロドプシン類タンパク質 を探すため、塩湖から好塩菌を単離して、16S rRNA 遺伝子配列解析より新種で あることを明らかにし、Halorubrum sp.ejinoor (He)と名付けた。He からゲノ ム DNA を抽出し、全ゲノム解析を行ったところ、三種類の細菌ロドプシン類タ ンパク質遺伝子、BR-like(HeAR)、HR-like(HeHR)、SRII-like(HeSRII)と 信号伝達タンパク質遺伝子(HeHtrII)を見つけた。第二章では、He の持つロド プシン類タンパク質遺伝子の mRNA 発現量やそれらの光駆動ポンプ活性、光反応 中間体について検討した。
24
2.1 好塩菌の単離
高度好塩菌は塩湖や塩田など高塩環境で生育するバクテリアで、古細菌の主 なグループの一つである。本研究では、中国内モンゴルエジノル塩湖 (図 2.1) から高度好塩菌を単離することにした。 図 2.1: 上図、中国内モンゴル自治区、新疆とチンハイ(google マップ出典)。 赤 A マークは試料を採集したエジノル塩湖である。下図、2012 年6月試料を 採集時のエジノル塩湖の写真。25
2.1.1 方法
2.1.1.1 試料
試料は中国の内モンゴルシリンゴル盟東ウジムチン旗エジノル塩湖から採取 した。エジノル塩湖の湖水を採取し、0.45 μM のフィルターろ過により菌を集 めた。2.1.1.2 菌の培養
集めた菌を Basal Salt (表 2.1)に懸濁して、好塩菌寒天培地に培養した。(37℃、 15 日、光照射あり)出た一個のコロニーを選んで新しい好塩菌培地 (表 2.2)が 入っているプレート中に広げた。この操作を繰り返し2回行い、好塩菌を単離 した。26
2.1.2 結果
菌の単離
集めた菌をプレートに培養すると図 2.2 で示しているように赤コロニーが出 た。離れている三つのコロニー(L1, L2, L3)を選んで 16S rRNA による同定を 行った。 図 2.2 : プレート上の好塩菌のコロニー。それらのコロニーか ら L1, L2, L3 のコロニーを選んで 16S rRNA による同定を行った。27
2.2 16S rRNA 遺伝子配列による菌の同定
2.2.1 方法
単離されたコロニーからゲノムDNAを抽出し、アーキア好塩菌の16S rRNAに特 異的なプライマーでPCRをかけ、増幅した配列を電気泳動にかけた。ゲルからDNA を抽出して、ライゲーションよりpT7 blueベクターにクローニングした。クロ ーニングした配列がベクターに入っているかどうかを確認するため、インサー トチェックを行った。目的遺伝子配列が入っているベクターを持つコロニーを 培養し、プラスミドDNAを取った。最後にシーケンスをして配列を決定した。得 られた配列とほかのアーキア好塩菌の16S rRNA遺伝子配列とを比較し分子系統 樹解析を行い、菌を同定した。2.2.1.1 ゲノム DNA 抽出
単離された三つのコロニーを各々3 ml の好塩菌液体培地 (pH 7.4) に入れて 培養した(37℃、光照射)。OD600 が 1.0 になるまで培養して、菌を遠心 (6,000g) により集めた。集めた菌を液体窒素で凍らせて、乳鉢で粉末にした。菌の重量を計って RSB バッファ(10mM Tris-HCl, 10mM NaCl, 25mM EDTA, pH 7.4 )を
2 ml/g 菌の割合で加えた。その後、10%の SDS 溶液を終濃度が 1%になるよう
に添加した。また、Proteinase K の粉末を最終濃度が 1 mg/ml になるように加
えて、37℃で 2 時間ゆっくりと(50 rpm)振蕩した。次に、全液量の 1/10 量の
5M NaCl と PCI 溶液 (50% phenol, 48% CCl4, 2% isoamyl)を加えた。軽く
遠心して、上層を取り、2 倍量のジエチルエーテルを加えてよく混ぜた。混ぜて、
28 を析出させ、滅菌したガラスロッドで巻き取った。取った DNA を繰り返して二 回エタノール沈殿を行い、ゲノム DNA とした。
2.2.1.2 PCR
得られたゲノム DNA を用いて、16S rRNA に特異的なプライマーで PCR を行っ た[37]。PCR で増幅した遺伝子配列を 2%のアガロースゲルで電気泳動を行った。 プライマー: P0023: 5 TTCCGGTTGATCCTGCC 3 P1525: 5 AAGGAGGTGATCCAGCC 3 PCR 反応 熱変性: 95 ℃ 3 分 熱変性: 95 ℃ 1 分 アニール: 50 ℃ 1 分 伸長反応: 72 ℃ 1 分 :2 へ戻って 30 サイクル行う。 伸長反応: 72 ℃ 10 分 4℃ keep29
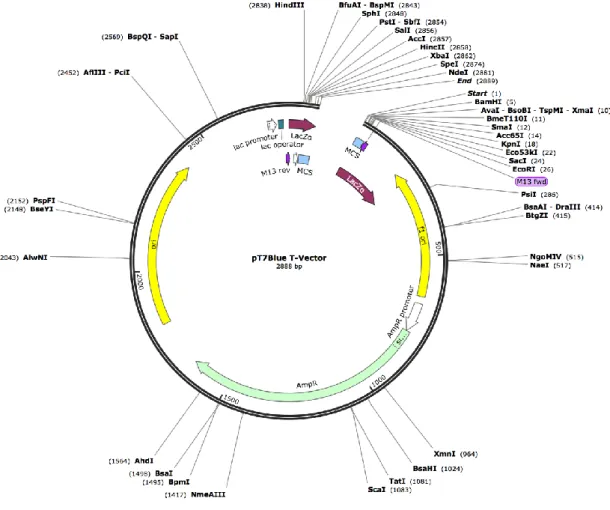
図 2.3 : pT7 blue T ベクター模式図。全長は 2888bp であり、黒 い線より制限酵素サイドを示している。
30
2.2.1.3 目的遺伝子配列をベクターへサブコロニング
PCR より得られた目的サイズの DNA バンドをゲルから回収した。回収した DNA を pT7 blue T ベクターへ導入した (図 2.3)。次に、トランスフォーメーション を行い、大腸菌を LB/Amp 寒天培地プレートにまいて 37℃で培養した。インサー トチェック PCR を行い、DNA の導入を確認した。 LB 培地(1L): Bacto Trypton 10g Bacto Yeastextract 5g NaCl 5g 1N NaOH 1mL --- dH2O up to 1L オートクレーブ滅菌 インサートチェック用プライマー: P7: 5 CGCCAGGGTTTTCCCAGTCACGAC 3 P8: 5 AGCGGATAACAATTTCACACAGGAAAC 3 PCR 反応 熱変性: 95 ℃ 3 分 熱変性: 95 ℃ 1 分 アニール: 50 ℃ 1 分 伸長反応: 72 ℃ 1 分 :2 へ戻って 30 サイクル行う。 伸長反応: 72 ℃ 10 分 4℃ keep31
2.2.1.4 プラスミド DNA 抽出
インサートチェックで確かめたコロニーを 3 ml の LB/Amp 液体培地に入れて、
37℃で 150 rpm、18 時間振蕩培養した。培養した菌を遠心して、MagExtractor-
Plasmid キット(ToYoBo, Osaka, Japan)を用いてプラスミド DNA を抽出した。
2.2.1.5 DNA 配列決定
得られたプラスミド DNA の濃度を調整して、BigDye® Teminator v3.1 cycle
sequencing kit を用いてシーケンス PCR を行った。BigDye® XTeminator 精製キ
ットを用いてサンプル DNA を調整した。最後に、Applied Biosystems 3130/3130xl
(Applied Biosystems, Foster City, USA)で 16S rRNA 遺伝子配列を決定した。
決定した DNA 配列を National Center for Biotechnology Information, NCBI
データベースに対して Blast 解析を行い、相同な配列を検出した。分子系統樹
は遺伝子配列解析ソフト GENETYX ver. 10.0.2 (Genetyx, Tokyo, Japan)を用い
て Neighbour-joining[38]法で作製した。 PCR 反応: 熱変性: 96℃ 1 分 熱変性: 96℃ 10 秒 アニール: 50℃ 5 秒 :2 へ戻って 30 サイクル行う。 伸長反応: 60℃ 4 分 4℃ keep
32
2.2.2 結果
16S rRNA に対する PCR
図 2.4 は各コロニーの PCR 産物を電気泳動した結果である。左のレーンから マーカー、コロニー(L1)、コロニー(L2)、コロニー(L3)である。写真か ら、試料から目的サイズの DNA 断片が増幅されていることが分かる。 図 2.4 : PCR 産物の電気泳動。M, マーカー;L1, L2, L3 はそ れぞれ単離された菌に対する PCR 結果である。L1, L2, L3 のコ ロニーを選んで 16S rRNA 遺伝子を増幅した。33
シーケンスより決定した 16S rRNA 遺伝子配列
シーケンスを行った結果 (図 2.5)、得られたコロニーL1、L2、L3 の 16S rDNA 遺伝子の配列は同一であった。つまり、三つの菌が一種類の菌という ことが分かった。 図 2.5 : 得られた 16S rRNA 遺伝子の配列34
16S rRNA による分子系統樹解析
GENETIX Ver.10: Phylogram Tree
Method: Neighbour-joining
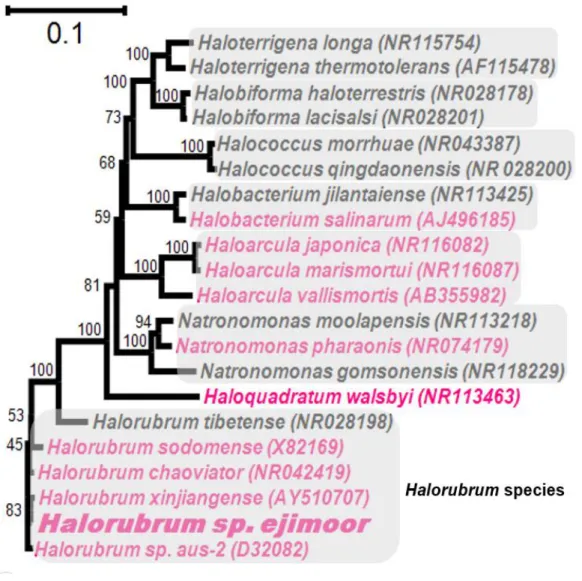
単離した好塩菌の 16S rRNA 遺伝子の配列と他のアーキア好塩菌のリボソーム
RNA 遺伝子配列と比較したところ Halorubrum 属菌と最も相同性が高かったが、
これまでに報告のない新規な菌であることがわかった (図 2.6)。そのため、
Halorubrum sp. ejinoor (He)と名付けた。
35
2.3
Halorubrum
sp. ejinoor か ら の ロ ド プ シ ン 類 タ ン パ
ク質の探索
同定された菌 He からゲノム DNA を抽出して BR に特異的なプライマーで PCR
を行い、シーケンスより配列決定して、National Center for Biotechnology
Information, NCBI データベースで Blast X 解析を行い、相同性の高い遺伝子
配列を探した。BR-like 遺伝子断片が得られたため、名古屋大学の井原先生に頼 み、ゲノム解析を行い、ロドプシン類タンパク質遺伝子を探索した。
2.3.1 方法
2.3.1.1 PCR
He のゲノム DNA に対して、BR に特異的なプライマーで PCR を行った。PCR 産 物を電気泳動で流して、そのサイズを確認した。増幅した配列をサブクローニングし配列を決定した。決定した DNA 配列を NCBI データベースに対して Blast
X 解析を行い、相同な配列を検出した。
2.3.1.2 ゲノム DNA 解析
He からゲノム DNA を抽出した。そのゲノム DNA を名古屋大学井原先生に送付
36
2.3.2 結果
BR の断片配列による PCR He のゲノム DNA を用いて BR に特異的なプライマーで PCR を行った結果、500bp ぐらいのところに目立ったバンドが出た (図 2.7)。これらのバンドが設計した プライマーより予測される BR の断片サイズと一致するため、バンドをゲルから 回収して、シーケンスを行い、配列を決定した。NCBI データベースからアミノ 酸レベルで相同性の高い配列を検索すると、BR-like ロドプシンタンパク質遺伝 子であることを分かった。 図 2.7 : Heからの BR 断片による PCR 解析。M, マーカー;L1, L2, L3 はそれぞれのコロニーによる PCR 解析。37
ロドプシン類タンパク質遺伝子分子系統樹解析
ゲノム解析を行った結果、三種類のロドプシン類タンパク質遺伝子 BR-like、
HR-like、SRII-like と光情報伝達タンパク質 HtrII を見つけた (図2.9)。そ
れらの全長アミノ酸は BR-like : 258, HR-like : 290, SRII-like : 256 であ
った。ロドプシン類タンパク質を分子系統樹解析から四つのグループ (BR, プ
ロトンポンプ;HR、クロライドイオンポンプ;センサリーロドプシン I;センサ
リーロドプシン II)に分けた (図 2.8)。我々の得た BR-like 遺伝子はHalorubrum
属 菌 か ら 見 っ け た ア ー キ ロ ド プ シ ン (AR) の グ ル ー プ に 入 り 、Halorubrum
xinjiangense の持つ AR (HxAR)[36]と相同性が 95%であり、最も高かった。BR
との相同性は 57%であった。得られた BR-like 遺伝子は AR 遺伝子との相同性が
高かったため、HeAR と名付けた。HeAR のアミノ酸配列からプロトンポンプ機能
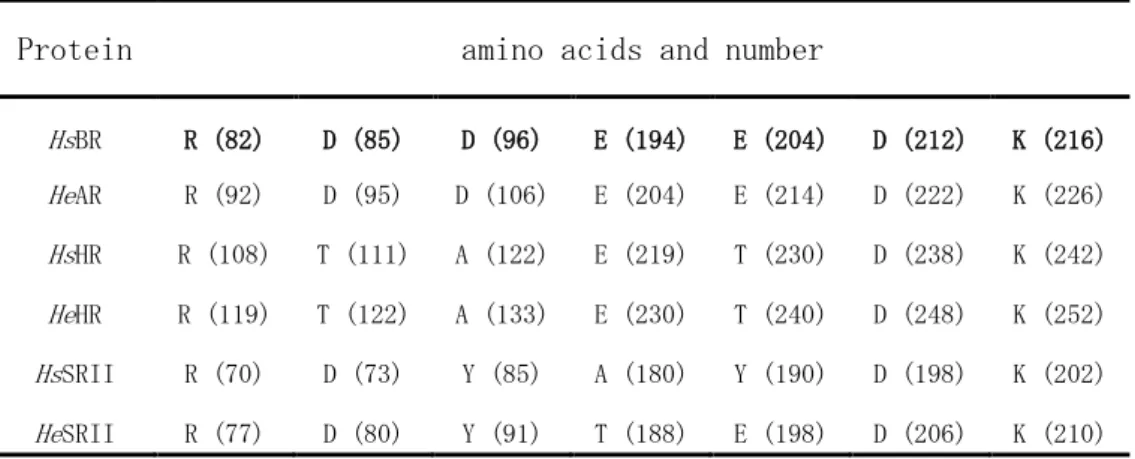
に重要なアミノ酸 Asp95 (BR:Asp85)、Asp106 (BR:Asp96)、Arg92 (BR:82)、Glu214
(BR:Glu204)と Asp222 (BR:Asp212)は保存されていることが分かった。得られた
HR-like (HeHR)遺伝子は Halorubrum sodomense[39]のもつ HR と相同性が最も高
く、84%であった。HR と比較して、クロライドイオンポンプ機能に重要なアミ
ノ酸 (表 2.3)は保存されていることが分かった (図 2.9)。SRII-like (HeSRII)
遺伝子はHalorubrum chaoviator の持つ SRII と相同性が最も高く、80%であり、
SRII とは 60%であった。分子系統樹解析に使われたロドプシン類タンパク質遺
38
表 2.3 He と Hs の持つ重要なアミノ酸配列比較
Protein amino acids and number
HsBR R (82) D (85) D (96) E (194) E (204) D (212) K (216) HeAR R (92) D (95) D (106) E (204) E (214) D (222) K (226) HsHR R (108) T (111) A (122) E (219) T (230) D (238) K (242) HeHR R (119) T (122) A (133) E (230) T (240) D (248) K (252) HsSRII R (70) D (73) Y (85) A (180) Y (190) D (198) K (202) HeSRII R (77) D (80) Y (91) T (188) E (198) D (206) K (210) 表 2.4 古細菌の持つロドプシン類タンパク質遺伝子 Accession number
Source organism BR family HR family SRI family
SRII family
Halobacterium salinarum M11720 2JAFA X51682 U62676
Haloarcula marismortui Q5UXY6 Q5V1N0 Q5UXM9 Q5V5V3
Q5V0R5
Haloarcula vallismortis BAA06680 D31881 D83748 Z35308
Haloarcula japonica BAA81816 WP_004591497* EMA29974 ND
Haloquadratum walsbyi Q18DH8 CAJ53165* ND ND
Q18DH5
Natronomonas pharaonis ND P15647 ND Z35086
Halorubrum chaoviator P69051 ND (P) BAB86796*
Halorubrum sp aus 2 P29563 ND ND ND
Halorubrum sodomense P96787 O93742* O93743* ND
Halorubrum xinjiangense AAS15567 ND ND ND
Halobacterium halobium XZ515 AF306937 ND ND ND
ND: not determined, (p): partial sequence, *: the physiological properties of the protein was not reported.
39
図 2.8 : マイクロバイアルロドプシン類タンパク質の分子系統樹解析。四つの クループ BR, プロトンポンプ;HR, クロライドイオンポンプと SRI, SRII, 光
センサーグルプに分けられた。 ‘Hc’, ‘Ha’, ‘Hrs’, Hh’, ‘Hx’,
‘Hs’, ‘Hw I (and Hw II)’, ‘Hm I (and Hm II)’, ‘Hj’, ‘Hv’ と ‘Np’ は Halorubrum chaoviator, Halorubrum sp. aus 2, Halorubrum sodomense, Halobacterium halobium XZ515, Halorubrum xinjiangense,
Halobacterium salinarum, Haloquadratum walsbyi, Haloarcula marismortui,
Haloarcula japonica, Haloarcula vallismortis, と Natronomonas pharaonis
40
図 2.9 : HeとHsの持つロドプシン類タンパク質のアミノ酸配列アライメント。
赤線は BR による α ヘリックス領域を示している。数字は BR のプロトンポンプ機 能に重要であるアミノ酸を示している。
41
2.4 RT-PCR 解析と Quantitative Real Time-PCR 遺伝子発現解析
He よりすべての RNA を抽出し、プライマーDNA、逆転写酵素により cDNA を合
成した。この cDNA をテンプレートにして PCR を行った。また、菌体内のロドプ
シン類を合成している mRNA の量比を知るため、Quantitative Real Time-PCR を
行った。使用したプライマーは He 菌より得た BR-like, HR-like, SRII-like,
HtrII-like 遺伝子に特異的なプラィマーである。
2.4.1 方法
2.4.1.1 RNA 抽出
He コロニーを取り、液体好塩菌培地に入れ、37℃で OD600 が 1 になるまで振
蕩培養した。 培養した菌を遠心より落として集めた。RNeasy Mini Kit (Qiagen,
Tokyo, Japan)を用いてHe からすべての RNA を抽出した。その後、DNase I を入
れて、混在している DNA を分解させた。
2.4.1.2 RT-PCR と Quantitative Real Time-PCR
He からの RNA をテンプレートとして、RT-PCR と Quantitative Real Time-PCR
を行った。He がロドプシン類タンパク質 mRNA を合成しているかどうかを確認す
るため、He のもつロドプシン類遺伝子に特異的なプライマー (表 2.5)で RT-PCR
42
RT-PCR 反応
Quantitative Real Time-PCR は One Step SYBR PrimeScriptTM PLUS RT-PCR Kit
(TaKaRa, Tokyo, Japan)を用いて StepOne-PlusTM Analyzer (Applied Biosystems,
Foster City, USA)より解析を行った。
43
2.4.2 結果
RT-PCR と Quantitative Real Time-PCR
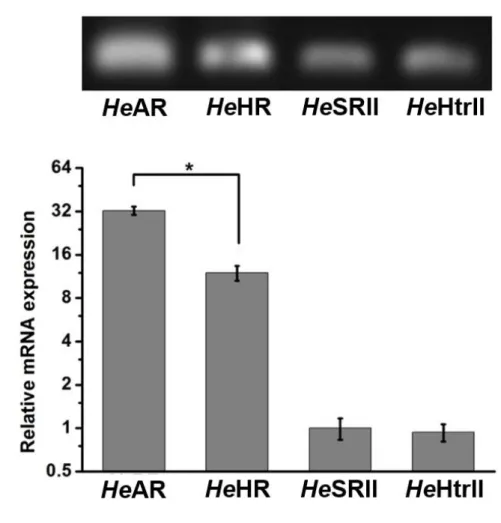
RT-PCR 解析からロドプシン類HeAR, HeHR, HeSRII と HeHtrII に対応するバン
ドが得られた (図 2.10a)。つまり、それらのタンパク質を合成する mRNA を検出
できた。Quantitative Real Time-PCR 解析結果よりそれらのタンパク質を合成
している mRNA の発現レベルが違うことが分かった(図 2.10b)。Hs の場合は菌体 膜中のロドプシン類タンパク質の量比が BR:HR:SRI:SRII= 1000:100:10:1 と報 告されている[14]。それに対して、我々の単離した He 菌のそれらの mRNA の発 現量比は HeAR:HeHR:HeSRII:HeHtrII= 30:10:1:1 であった。我々は菌体内の mRNA の量比を明らかにしたが、タンパク質の発現量は調べなかった。Hs の場合、 BR と HR タンパク質の発現量は菌体液の酸素や光量によりコントロールすること が可能と報告されている[40, 41]。He については今の段階では分からない。
2.5 RT-PCR と Quantitative Real Time-PCR 用プライマー
Halorubrum sp. ejinoor
Forward primer Reverse primer
HeAR 5` AGACGCTCTGGTTGGGTATAGG 3` 5` TACTCGCGAGCCTCCTTGTC 3`
HeHR 5` GAAATGACGCAGACGGAGATG 3` 5` GTTCCGTCCCATGTAGACGAA 3`
HeSRII 5` AACATCACCGTGGTGCTGTG 3` 5` TTCGAGACGATGTCGAGGTAGAC 3`
44
図 2.10 : Heの持つロドプシン類タンパク質と光情報伝達タンパク質を合成
する mRNA の発現量の比較。(a) それぞれロドプシン類タンパク質と光情報 伝達遺伝子 RT-PCR 解析。(b) Quantitative Real-Time RT-PCR 解析より HeAR,
HeHR, HeSRII と HeHtrII 遺伝子の発現量比較。*は有意差 P<0.02 を示し
45
2.5 菌体膜小胞の光駆動イオンポンプ活性
エジノル塩湖から単離された菌He がイオンポンとして働く AR-like, HR-like 遺伝子を持つことが分かったため、それぞれのイオンポンプ活性を調べるため 菌体膜小胞を作成して、イオンポンプ機能を測定した。2.5.1 方法
2.5.1.1 菌体膜小胞作製
He コロニーを液体好塩菌培地に培養して、遠心より菌を集め、菌体膜から凍 結融解法[42]により菌体膜小胞を作製し、その光駆動ポンプ活性を調べた。2.5.1.2 光照射に伴うプロトン濃度変化の測定
作製した菌体膜小胞を 3M NaNO3と 4M NaCl 溶液に懸濁してテンプレートとし てイオンポンプ実験に使用した。テンプレート溶液の菌体膜小胞のロドプシン の量を同一にした。それらに強い光を当てて、外液のプロトンの濃度を pH メー タで測った。また、CCCP と TPP+ 存在条件で光を当てて外液のプロトン濃度変 化を測った[43]。46
2.5.2 結果
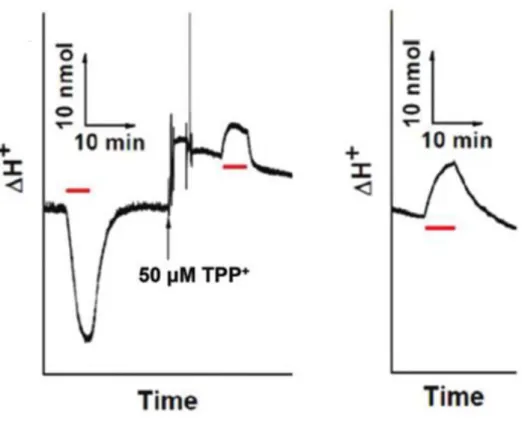
菌体膜小胞イオンポンプ活性
図 2.11 に菌体膜小胞を 4M NaCl 溶液に懸濁してイオンポンプ活性を測った 結果を示している。試料に光を当てて測定すると外液のアルカリ化が見られた (図 2.11)。この結果は HR のイオンポンプ活性と似ている。HR は内向きクロラ イドイオンポンプであるため、光を当てると、クロライドイオンを細胞内側へ 運び、それに応じて膜を介した電気化学ポテンシャルができ、外液のプロトン がその電気化学ポテンシャルをキャンセルするため、細胞中へ流入する。その 結果、外液のプロトン濃度が下がり、外液のアルカリ化が見られる[6]。その後 に試料液に CCCP を加えてから光を当てて測定すると CCCP を入れていない試料 液のプロトン変化より変化は大きくなり、外液の酸性化が見られた。CCCP はプ ロトンのイオノフォアである。CCCP を菌体膜小胞液に入れるとプロトンは、細 胞膜を自由に行き来できるようになる[44]。そのため、細胞膜を隔てたプロト ンの電気化学ポテンシャル勾配を打ち消してしまい、HR の活性が強く見られる ようになった。以上の結果より、我々の作製した膜小胞は内向クロライドイオ ンポンプ持つことが分かった。 光駆動プロトンポンプ活性を見出すため、TPP+を試料液に加えて光を当てて 測定すると外液の酸性化を見だした (図 2.12 左)。TPP+は脂溶性のカチオンで ある。膜を透過できるので、クロライドイオンポンプ活性によって受動的に移 動するプロトンの代わりに TPP+が細胞中へ入ってくる[44, 45]。その結果、内 向きクロライドイオンポンプ活性をキャンセルしたため、外向きプロトンポン プの活性が見出された。このプロトンポンプ活性がHeAR に由来することを明ら かにするため、作製した菌体膜小胞を 3M NaNO3に懸濁して、クロライドイオン47 がない条件でのイオンポンプ活性を測定した。その結果、外液の酸性化が見ら れた。つまり、クロライドイオンがない溶液では HR は内向きクロライドイオン ポンプとして働けないため、外液のアルカリ化が見られない。しかし、AR や BR はクロライドイオンに関係しないため、プロトンポンプ活性は影響を受けない。 以上の結果より、作製した菌体膜小胞に HeAR と HeHR が含まれていて、それぞ れ外向きプロトンポンプと内向きクロライドイオンポンプとして働くことを明 らかにした。 図 2.11 : 菌体膜小胞イオンポンプ活性の測定。赤バーは光 (590 ± 8.5 nm) を照射した時間である。菌体膜小胞を 4M NaCl 溶液に懸濁して、光を当てて測 定すると、プロトンの濃度が下がり、光を切ると、また、平衡ラインに戻った。 その後、10 μM CCCP を加えて、もう一回光を当てて測定を行った。
48
図 2.12 :左図、菌体膜小胞を 4M NaCl 溶液に懸濁して、測定を行った。光を
当てるとプロトンの濃度が下がり、また、平衡ラインに戻った後に 50 μM TPP+
を加えて、測定を行った。右図、菌体膜小胞を 3M NaNO3溶液に懸濁して、光
49
2.6 菌体膜の光反応
三種類のロドプシン類タンパク質遺伝子を得られので、それらの光反応特性 を調べるため、Hs の紫膜を分離する方法と同じように He 菌体膜を分離して、レ ーザー誘起吸光度変化より光反応中間体を検討した。2.6.1 方法
2.6.1.1 菌体膜の分離
内モンゴル塩湖から単離した菌 He と Hs を同時に好塩菌培地プレートに培養 した。He と Hs のコロニーを 3ml の液体好塩菌培地に培養した(7 日間、37 度の 恒温槽、光照射、100rpm)。その培養液から 2ml 取って 35ml 培地が入っている 100ml の三角フラスコに入れて培養した(7 日間、37 度の恒温箱、光照射、100rpm)。培養した菌を遠心(6,000g)により集め、Basal Salt に懸濁して、10mM Tris-HCl
pH 7.5 バッファで透析を行った。その後、透析液を遠心 (50.000g)して、膜
懸濁液を回収した。次に、60~30%のショ糖溶液のステップ勾配で超遠心を行
い、菌体膜の分離を試みた。
2.6.1.2 レーザー誘起吸光度変化の比較
得られた菌体膜のレーザー誘起吸光度変化を光励起 Nd:YAG laser pulse
(Nd:YAG, 532 nm, 7 ns, 5 mJ per pulse)で観測した[43, 46]。菌体膜をそれ
ぞれ 50 mM HEPES, 1M NaCl バッファと 1M Na2SO4 溶液に懸濁して波長 410 nm, 500
50
2.6.2 結果
菌体膜分離と吸収スぺクトル
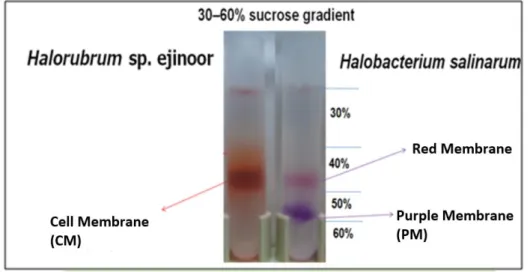
培養した He と Hs を遠心により落として集めるとそれぞれ赤色と紫色をして いた (図 2.13)。内モンゴル塩湖の写真をみると赤色の塩湖であることを分かる。 おそらく好塩菌の色から湖が赤になったと考えられた。 Hs の紫膜(PM)と赤膜はショ糖密度勾配遠心よりきれいに分離された(図 2.14 右)。しかし、He の膜は Hs の膜のようにきれいに分離できなかった (図 2.14左)。分離できなかった膜を Cell membrane (HeCM)と名付けた。それらの吸収ス
ペクトルを測定するとHsPM では 570 nm に極大吸収波長を持ち、これが BR の吸 収を示している (図 2.15)。HeBM の吸収スペクトルにはカロテノイドのピー クが見られた。つまり、HeCM には大量のカロテノイドが含まれていることが分 かる(図 2.15)。Hs の菌体膜では PM はバッチ状の特異的な構造を持ち、BR 二 次元結晶構造を取っているため、ショ糖密度勾配遠心より紫膜と赤膜を分離す 図 2.13 : 菌体色の比較。左図は Halorubrum sp. ejinoor 菌色である。右図は Halobacterium salinarum 菌色である。
51
ることができる[2, 47, 48]。しかし、He の膜をショ糖密度勾配遠心より分離で
きなかったため、Hs の紫膜のように特異的な構造を取っていないと考えられた。
図 2.14 : Halorubrum sp. ejinoor とHalobacterium salinarumのショド密
度勾配遠心より分離。左図は Halorubrum sp. ejinoor 菌体膜の分離を示し
ている。その膜を Cell membrane (CM)と名付けた。右図は Halobacterium
salinarum 菌体膜の分離を示している。分離した膜をそれぞれ Red membrane (RM) と Purple membrane (PM)と名付けた。
図 2.15 : CM と PM の吸収スぺクトル。試料を 50 mM HEPES, pH 7.0 で調整
52
菌体膜光反応
BR と HR が光を受容するとレチナールが all-trans から 13-cis に異性化して、 いくつかの中間体を経て元に戻る光化学反応サイクルを行う。BR の場合、K, L, M, N, O 中間体を持ち、最後に元の BR に戻る[49, 50]。HR の場合は L1, L2, N, O, HR` 中間体を経て元の HR に戻る[9, 51]。光反応中間体はそれぞれの特徴的 な吸収極大をもっている (図 1.6)。 特に、重要なのは M 中間体がただ BR の光サイクルに存在していて、HR にはな いことである。HsPM のレーザー誘起吸光度変化から BR を示す波長 570 nm の吸 光度が減るのに対応して、M 中間体を示す 410 nm の吸光度が増加している。つ まり、BR の光反応が始まって、BR から M 中間体が生成していることが分かる。 そのあと、M 中間体が減ると共に、O 中間体を示す 670 nm の吸光度が増加して いる。同時に BR を示す 570 nm の吸光度も増加している。つまり、M が、次の O 中間体に変化していることがわかる。最後に M と O 中間体が完全になくなると、 BR も完全に元に戻った (図 2.16c)。このようにHeCM の結果から M 中間体を示 す 410 nm の吸光度の変化が BR の M 中間体と同様に出ていることが分かる。つ まり、この M 中間体の変化はHeAR 由来の中間体と考えられた。 HR では L と O 中間体はクロライドイオン濃度依存的に平衡していると報告さ れている[43]。つまり、クロライドイオン増加に伴い、平衡が N へ移動、逆に クロライドイオンを減らすことで N 中間体も減っていくことが分かっている。 HeCM 菌体膜試料をクロライドのある条件 (図 2.16a)とない条件で (図 2.16b) 500 nm の吸光度を比較したところ、大きな吸光度変化の違いを見られた。この 現象から、HeHR 由来の L 中間体の吸光度変化を示していて、分離できなかった HeCM 膜に HeHR も存在していると考えられた。53
O 中間体を示す 670 nm の吸光度変化も見られた。しかし、BR と HR にも O 中
間体が存在するため、この結果からHeAR 由来か、HeHR に由来する O 中間体かを
判断できない。以上の結果より、HeCM に HeAR と HeHR が含まれ、それぞれの光
反応サイクルを行うことができていることを明らかにした。 図 2.16 : HeCM とHsPM のレーザー誘起吸光度変化の比較。HeBM 試料をクロライ ドイオンがあるとない条件で四つの波長、570, 410, 500, と 670 nm で測定を行 った。(a) HeCM を 50 mM HEPES, 1 M Na2SO4 ( Cl−なし) に溶解して測定を行った。 (b) HeCM を 50 mM HEPES, 1 M NaCl ( Cl−あり) に溶解して測定を行った。(c) HsPM を 50 mM HEPES, 1 M NaCl ( Cl−あり) に溶解して四つの波長、570, 410, 500, と 670 nm で測定を行った。
54
2.7 まとめ
我々は内モンゴルエジノル塩湖から好塩菌の 16S rRNA 配列の同定を行い、新
規な Halorubrum 属菌を単離して Halorubrum sp. ejinoor と名付けた。三種類
のマイクロバイアルロドプシン類タンパク質遺伝子(HeAR, HeHR, HeSRII)を
Halorubrum sp. ejinoor から見つけた。
RT-PCR と Quantitative RT-PCR 解析より、菌体内にそれらのタンパク質を合
成する mRNA が発現していて、それらの比率がHeAR : HeHR: HeSRII = 30 : 10 :
1 であることを明らかにした。
菌体膜小胞の光駆動イオンポンプ活性よりHeAR 由来のプロトンポンプ活性と
HeHR 由来のクロライドイオンポンプ活性を見いだした。つまり、菌体膜小胞に
はHeAR と HeHR が含まれていると考えられた。
HeCM の光反応サイクルより BR(HeAR)の M-like 中間体と HeHR 由来の N/L 中
間体の変化を見出した。つまり、HeCM に HeAR と HeHR がふくまれ、それぞれの
光反応サイクルを行っていることを明らかにした。
55
第三章
Halorubrum
sp.ejinoor の持つアーキ
ロドプシンの大腸菌による発現精製
He から三種類のロドプシン類タンパク質遺伝子を見つけた。He の菌体膜では HeAR と HeHR はイオンポンプとして機能していることを明らかにした。また、光 反応も行っていることが分かった。それらのポンプ機能や光反応サイクルを詳 しく解明するためには、それぞれを発現精製する必要がある。そのため、第三 章では得られた三種類のロドプシン類タンパク質を大腸菌による発現精製を行 い、それらのポンプ機能と光反応について検討した。56
3.1 アーキロドプシンの発現精製
発現精製するロドプシン類タンパク質遺伝子を発現ベクターpET21c(+)へ導 入して、C 末端に 6×His がついている発現ベクターを作製した[43]。その後に 作製したベクターを持つ大腸菌 BL21(DE)を培養して、IPTG やレチナールにより 目的ロドプシン類タンパク質を発現させる。発現させたタンパク質を界面活性 剤 n-Dodecyl-β-D-maltoside (DDM)で可溶化させて、Ni アフィニテイカラムに より精製を行った[52]。3.1.1 方法
3.1.1.1 発現ベクターの作製
pET-21c(+)を用いて発現ベクターを作製した。pET ベクター発現システムは、培地への IPTG 添加によって T7 RNA ポリメラーゼを誘導し、T7 RNA ポリメラー
ゼ応答性プロモーターの下流にクローニングされた目的遺伝子を高収率で発現
させるタンパク質発現誘導システムである (図3.1)。ロドプシン類タンパク
質への発現にも広く使われている。我々のロドプシン配列を図3.1のように
57
図 3.1 : 発現ベクターへ導入したロドプシン類タンパク質遺伝子。
表 3.1 : He の持つロドプシン類タンパク質による発現プライマ
58
59
60
図 3.4 : His tag 付けたロドプシン類タンパク質のNi アフィニテイ
61
3.1.1.2 アーキロドプシン遺伝子配列の発現ベクターへ
の導入
pET-21c(+)ベクターの NdeI/XhoI サイド部位にロドプシン類タンパク質遺伝 子を導入することにした。そのため、まず、pT7 blue ベクターに入っているロ ドプシン類タンパク質遺伝子に対して制限酵素サイド配列がついているプライ マーで PCR を行う。増幅されたロドプシン類タンパク質遺伝子をシーケンスに より配列を確認した後、制限酵素 NdeI/XhoI サイドトで切断した pET-21c(+) ベクターへ導入した。導入した配列を発現プライマー (表 3.1)で PCR を行い、 出たバンドを回収してシーケンスによりロドプシンタンパク質遺伝子が発現ベ クターへ入ったかを確認した。3.1.1.3 大腸菌によるアーキロドプシンの発現
ロドプシン類タンパク質遺伝子が入っている発現ベクターを持つ大腸菌を液 体 LB/Amp 培地に入れて 37℃で培養する。OD600 が 0.5~0.6 になるまで培養し て、誘導物質 1mM IPTG と 10 μM レチナールを入れて、さらに6時間振蕩する (図 3.2)。次に、培養した菌を遠心により落として集める。集めた菌を Lysis バッ ファ (50 mM Tris-HCl pH 8.0)に懸濁してガラスビーズで菌体を破砕する。破 砕した液を軽く遠心してビーズを落とし、その上清を更に高速で遠心して沈殿 物質を回収して 1.5%の DDM を入っている 50 mM Tris-HCl pH 8.0 バッファで懸 濁して 4℃で 12 時間置き、膜タンパク質ロドプシン類を可溶化させる (図 3.3)。 最後に可溶化したロドプシン類タンパク質を Ni アフィニテイカラムにより精製 を行った (図 3.4)。62
3.3.1.1 発現アーキロドプシンの可溶化と Ni アフィニテ
イカラムによる精製
可溶化させた His タグ導入タンパク質溶液を Ni2+プレチャージの Ni-NTA ア
ガロース担体を使用して、カラムでの精製を行った。まず、平衡バッファ(50 mM
MES, 300mM NaCl, 50 mM imidazole, pH 6.5)で Ni-NTA 担体を洗い、その後に
可溶化しているタンパク質液を加えて、37℃で 2 時間置き、Ni-NTA 担体と His
タグ導入ロドプシン類タンパク質を結合させた。その後に洗浄バッファ (50 mM
MES, 300mM NaCl, 50 mM imidazole, pH 6.5)で 3 回ぐらい洗い、溶出バッフ
ァ(50 mM Tris-HCl, 300 mM NaCl, 300 mM imidazole, pH 7.5)を加えて、His
タグが付いているロドプシン類タンパク質を回収する。最後に透析を行い、イ
ミダゾールを除き、純度の高いロドプシン類タンパク質を得る。
3.1.2 結果
He
の持つロドプシン類タンパク質の発現精製
HeAR, HeHR, HeSRII 三種類のロドプシン類遺伝子を発現ベクターpET21c(+)
に導入して大腸菌により発現したところ、HeAR だけを発現精製することができ た。HeAR の発現過程では誘導物質 IPTG とレチナールを菌液に入れて6時間培養 すると菌液色が淡黄色から紫赤色になる(図 3.5)。精製したHeAR は紫色であっ た。 SDS-PAGE 解析の T+に 28KDa ぐらいに目立った太いバンドが見られた。このサ イズが E のレーンに出ているバンドのサイズと一致していることが分かる(図 3.6)。T0では T +のように目立ったバンドが見られなかった。発現ベクターへ導
63 析の結果と一致するため、HeAR を大腸菌より発現精製することができた。精製 したHeAR は BR と同様に極大波長を持ち、550 nm であった(図 3.4a)。 図 3.5 : HeAR 遺伝子を導入したベクターを持つ大腸菌と導入していないベク ターを持つ大腸菌の色を比較。(a) HeAR 遺伝子を導入していない大腸菌。(b) 誘導させたHeAR 遺伝子を持つ大腸菌。(c) 精製したHeAR。 図 3.6 : HeAR の発現精製 SDS-PAGE 解析。M, マーカー;T0, 誘導する前の 菌;T+, 誘導後の大腸菌;E, Ni カラム後のHeAR。
64
3.2 アーキロドプシンの吸収スペクトルと CD スペクトル
BR の場合、CD を測定すると、BR は紫膜中では三つロドプシン分子で三量 体を作っているため、その構造を反映して、強いレチナール同志の相互作用を 示す CD スペクトルが得られる(図 1.2)[53, 54]。大腸菌から精製したHeAR の構 造を検討するためHeAR の CD 測定を行った。
3.2.1 方法
CD の測定には大腸菌から精製したHeAR 溶液 (0.15% DDM, 50mM Tris-HCl, pH 7.0)と紫膜懸濁液 BR (50mM Tris-HCl, pH 7.0) が使用された。測定には JASCO・ J-2D が用いられた。3.2.2 結果
C・D スペクトル
HsPM の懸濁液(50mM Tris-HCl pH 7.0)がレチナール同志の相互作用を示す CD スペクトルを持っていることが分かる(図 3.7b)。精製したHeAR を 0.15%界面活 性剤 DDM のバッファ(50mM Tris-HCl pH 7.0)に溶解して測定した結果、BR のよ うにレチナール同志の相互作用を示す C・D スペクトルを得た。つまり、この二 次構造から界面活性剤が存在する条件で精製したHeAR は三量体構造を取ってい ることが分かった。HeAR と BR の吸収スぺクトルの極大波長が違うため、HeAR の C・D スペクトルが BR より 5 nm 程度短波長へ移動している(図 3.7b)。65
図 3.7 : 精製したHeAR と BR の吸収スペクトル (a)と CD スペクトル
(b)。赤,はHsPM (BR)であり、 黒はHeAR である。50mM Tris-HCl, 500mM NaCl, 0.15% DDM, pH 7.0 に溶解して測定した。
66
3.3 アーキロドプシンの明暗順応と発色団の解析
精製した HeAR は BR のように明暗順応型性質を持っているかどうかを調べる ため、HeAR に対して明暗順応と発色団レチナールの解析を HPLC 解析より検討し た。3.3.1 方法
3.3.1.1 明暗順応
精製したHeAR 溶液 (0.15% DDM, 50mM Tris-HCl pH 7.0)と紫膜懸濁液 BR (50mM Tris-HCl pH 7.0)を少なくとも一夜常温で暗所に放置したものを暗順応型HeARD と BRDとみなした。また、それらの試料を 500 nm の光で 10 分間以上照射したも のを明順応型HeARLと BRLとみなした。それらの試料の光当てた時間による吸収 スペクトルの変化を測定した。また、十分光を当てた後のスペクトルの戻りを 測定した。スペクトルの測定は MPS 2000 記録分光光度計(Shimadzu, Kyoto, Japan)で行った[55]。3.3.1.2 発色団レチナールの抽出および HPLC 解析
暗順応型のHeARDと明順応型のHeARL発色団レチナールの量を調べるため、高 速クロマートグラフを用いて解析を行った[56]。暗順応、または明順応させた試料HeAR と BR 溶液を 100 μl 取り、100 μl Hydroxylamine, 300 μl Methanol
を加えて、10 分程度よく混合させる。その後、600 μl Hexane を加えて、強
く振蕩後、遠心により液を二層分離する。上液を取り、N2 ガスで有機溶媒を飛
ばしてしまい、50 μl Hexane を加えたものを液クロ用試料とする。高速液体ク
67
3.3.2 結果
He
AR の明順応と暗順応
BR では明暗順応型が存在している。BR を暗所で一夜以上置いて吸収を測定 すると λmax は 558 nm であり、640 秒 500 nm の光を当てると、λmax は長波 長へ 10 nm 移動して 568 nm になる(図 3.8c)。得られた BR を暗所に 80 分ぐら い置くと元の短波長側に吸収極大を持つ BR に戻る特徴がある(図 3.8d)[56, 57]。
HeAR 試料 (0.15% DDM, 50mM Tris-HCl pH 7.0)を一夜暗放置作った HeARDは 550
nm に λmax をもつ。この試料HeARDを 500 nm の光で照射しHeARLを得た (図 3.8a)。
得られたHeARLを 20℃で暗放置して置くとおよそ 160 分でHeARDに変化した (図
3.8b)。HeAR の暗順応から明順応、また、明順応から暗順応への変化は BR の変
化より遅いことを分かった。この現象は、HeAR と BR の明暗順応型の持つレチナ
ールの異性体組成が違うために起こったと考えた。そのため、明暗順応型の BR
68 図 3.8 : 20℃でのHeAR とBRの明暗順応。(a) HeAR の暗順応から明 順応への吸収変化。HeARD (curve 1)に 500 nm の光を照射して HeARL (curve 11)を得た。照射時間は curve 2 から 11 まで各 10, 10, 20, 40, 80, 160, 320, 640, 1280, 2560 秒。 (b) 得たHeARL (curve 1)を暗 放置するとHeARD (curve 8)に変化する。Curve 2 から 8 まで暗保時間 は、5, 5, 10, 20, 40, 80, 160, 320 分。(c) BR の暗順応から明順 応への吸収変化。BRD (curve 1)に 500 nm の光を照射して BRL (curve 8)を得た。照射時間は curve 2 から 8 まで各 10, 10, 20, 40, 80, 160, 320 秒。 (b) 得た BRL (curve 1)を暗放置すると BRD (curve 7)に変 化する。Curve 2 から 7 まで暗保時間は、5, 5, 10, 20, 40, 80 分。