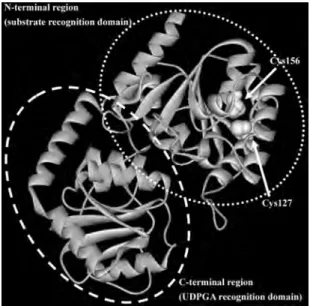
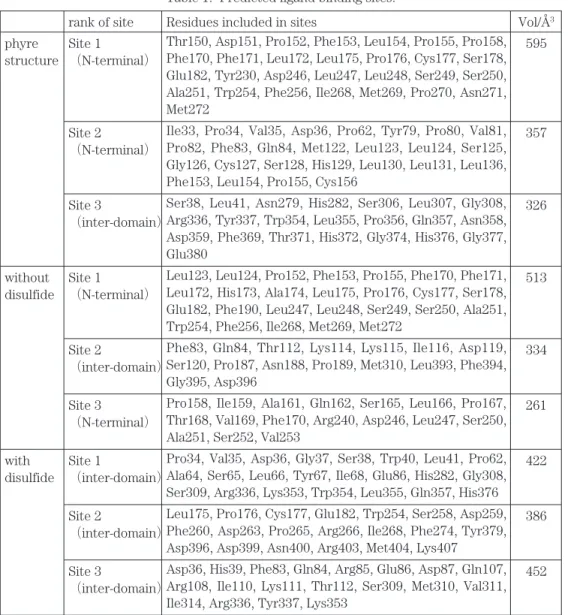
ウリジン二リン酸グルクロン酸転移酵素
6
0
0
全文
(2)
(3)
(4)
(5)
(6)
図
関連したドキュメント
上げ 5 が、他のものと大きく異なっていた。前 時代的ともいえる、国際ゴシック様式に戻るか
これらの定義でも分かるように, Impairment に関しては解剖学的または生理学的な異常 としてほぼ続一されているが, disability と
いメタボリックシンドロームや 2 型糖尿病への 有用性も期待される.ペマフィブラートは他の
ポンプの回転方向が逆である 回転部分が片当たりしている 回転部分に異物がかみ込んでいる
つの表が報告されているが︑その表題を示すと次のとおりである︒ 森秀雄 ︵北海道大学 ・当時︶によって発表されている ︒そこでは ︑五
と言っても、事例ごとに意味がかなり異なるのは、子どもの性格が異なることと同じである。その
注)○のあるものを使用すること。
次亜塩素酸ナトリウムは蓋を しないと揮発されて濃度が変 化することや、周囲への曝露 問題が生じます。作成濃度も