改良
VISCANA 新機能
説明書
Rev.1.0
i
目次
1. VISCANA 機能の改良 ... 1
1.1. VISCANA ウィンドウ外観 ... 1
1.2. フラグメント番号表示 ... 1
1.3. クラスタリング配列の選択機能 ... 2
1.4. データ読込・保持仕様の追加・修正 ... 3
1.5. CSV ファイル出力機能 ... 3
1.6. インストーラ作成 ... 5
1.7. 説明書作成 ... 5
2. ソフトウェア仕様... 6
2.1. 動作環境 ... 6
2.2. インストール... 6
2.3. 起動 ... 6
2.4. 操作 ... 6
3. 使用方法 ... 7
3.1. CPF ファイルの準備 ... 7
3.2. 配列データの取得 ... 7
3.3. 配列データの編集 ... 9
3.4. アラインメントデータの取得 ... 9
3.5. アラインメントデータの編集 ... 11
3.6. アラインメントデータの配置 ... 12
3.7. VISCANA 実行 ... 13
4. 課題 ... 17
1
1. VISCANA 機能の改良
BioStation Viewer Open1.0 rev10.2 に対して、相互作用エネルギー成分分割に係るク
ラスター解析可視化機能の改良を行い、
BioStation Viewer Open1.0 rev10.4 とした。以
下では、この機能改良の結果について述べる。
1.1. VISCANA ウィンドウ外観
改良の結果
VISCANA ウィンドウは、図 1 に示す外観となった。実施項目(1)~(4)の
関連箇所について併せて示す。これらの改良箇所について、以下に順次述べる。
図
1 VISCANA ウィンドウの外観と実施項目(1)~(4)との関連箇所
1.2. フラグメント番号表示
VISCANA のタイルに表示されるポップアップに、アラインメント後のフラグメント
番号を追加した(実施項目
(1))。併せて、番号の意味を示すための文字を併記するように
した。
改良後のポップアップの表示を図
2 に示す。ポップアップの表記で、“1L2J ETC600”
は各
Check Point File(以下 CPF)に記載されたラベルを、seq.=45 は複数の CPF 間で
共通となるアラインメント後の配列番号を、
frag.=42 は各 CPF 個別のフラグメント番
2
図
2 ポップアップ
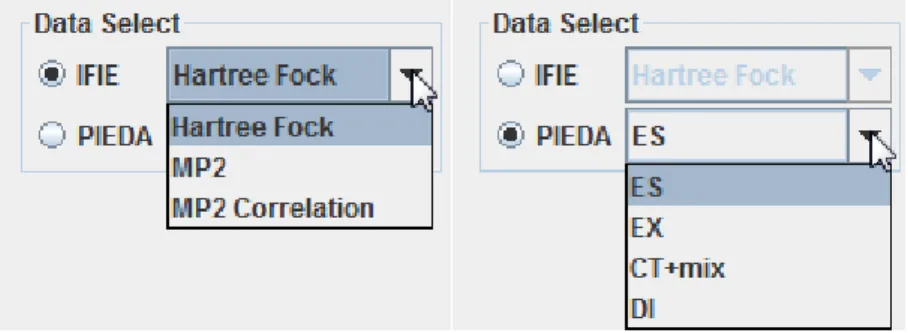
1.3. クラスタリング配列の選択機能
クラスタリングに用いる
IFIE の種類や PIEDA の成分を選択できるようにした(改良
項目
(2))。本改良に併せて、選択している成分を図の凡例に示すようにした。
改良によるクラスタリング配列の選択インターフェースを図
3 に、凡例への選択成分
の表示例を図
4 に示す。凡例のエネルギー範囲については、図 5 に示す中央下部にある
インターフェースにて最小値
(Min)、最大値(Max)をテキストにて設定するようになって
いる。今回の改良に合わせて、記入エリアの右側にプルダウンを設け、
10~100 までを
10 刻みにて、正負対称に設定できるようインターフェースを追加した。
図
3 クラスタリング配列の選択(左:IFIE、右:PIEDA)
図
4 クラスタリング配列の凡例への表示:IFIE, HF (Hartree Fock) の例
3
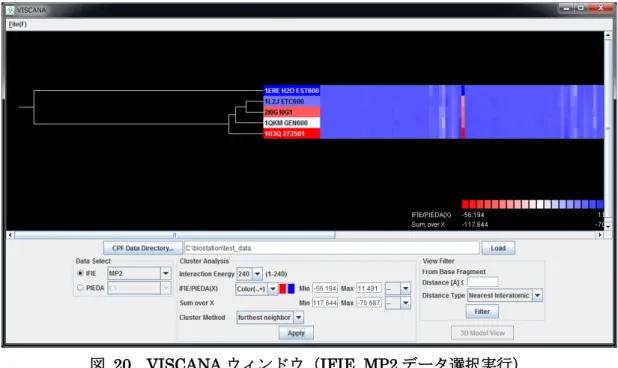
配列の選択によるクラスタリングの例について、図
6 に示す。上段では、IFIE の MP2
についてのクラスタリングを、下段では
PIEDA の ES についてのクラスタリングの状
況を示している。両者のクラスターの形成(ツリー構造)は異なっており、成分の違い
を反映している。
図
6 クラスタリング成分の選択による表示例(上:IFIE, MP2、下:PIEDA, ES)
1.4. データ読込・保持仕様の追加・修正
前項のクラスタリング配列の選択機能に必要となるデータ読込・保持機能を作成した
(実施項目
(3))。
CPF に含まれるフラグメント間相互作用エネルギーの各成分(IFIE の HF、MP2、
MP2 Correlation、PIEDA の ES、EX、CT、DI)の算出に必要なデータを読み込み、適
切な形式へと変換してプログラム実行時のメモリ上に保持できるようにしている。
図
3 のインターフェースを用いてこれらを切り替え、図 6 に示したように異なる成
分についてのクラスタリング結果の表示の切り替えが行えるようになっている。
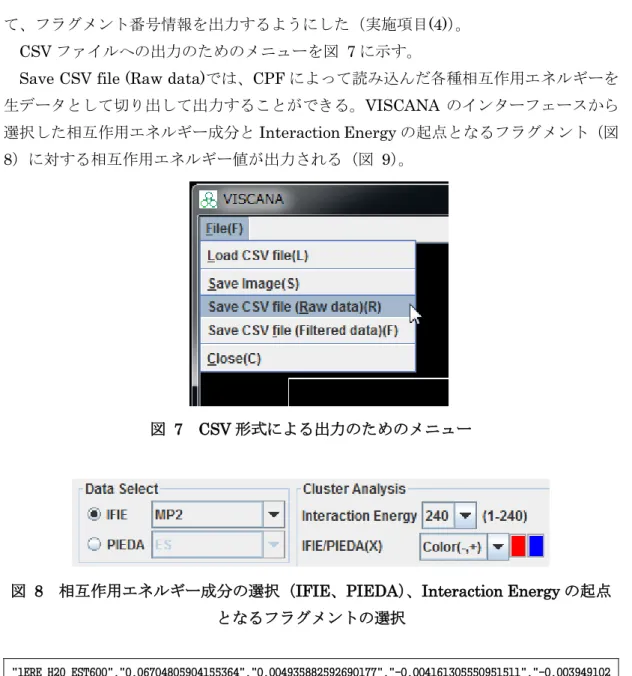
1.5. CSV ファイル出力機能
選択した
IFIE 種や PIEDA 成分の値を CSV ファイルに出力できるようにした。併せ
4
て、フラグメント番号情報を出力するようにした(実施項目
(4))。
CSV ファイルへの出力のためのメニューを図 7 に示す。
Save CSV file (Raw data)では、CPF によって読み込んだ各種相互作用エネルギーを
生データとして切り出して出力することができる。
VISCANA のインターフェースから
選択した相互作用エネルギー成分と
Interaction Energy の起点となるフラグメント(図
8)に対する相互作用エネルギー値が出力される(図 9)。
図
7 CSV 形式による出力のためのメニュー
図
8 相互作用エネルギー成分の選択(IFIE、PIEDA)、Interaction Energy の起点
となるフラグメントの選択
"1ERE H2O EST600","0.06704805904155364","0.004935882592690177","-0.004161305550951511","-0.003949102 "1L2J ETC600","0.09012984097353183","-0.01233636905089952","-0.06828176585258916","-0.01509068469749 "2I0G I0G1","0.017258612366276793","-0.001573896166519262","0.018046159180812538","0.007235390599817 "1QKM GEN600","0.06880561851721723","-0.021663096427801065","0.014909053323208354","0.00948922598036 "1U3Q 272501","0.03297848604415776","-0.022831991052953526","-0.029928279720479622","-0.030791792451
図
9 Save CSV file (Raw data)の出力
Save CSV file (Filtered data)メニューでは、さらに Interaction Energy の起点とな
るフラグメントからの距離によって選択(図
10)されたフラグメントに対する相互作用
エネルギーのみ(図
11)を出力することができる。この場合には、エネルギー値の一部
5
図
10 距離によるフィルタリング設定インターフェース
図
11 Filter によって選択された相互作用エネルギー
"1ERE H2O EST600","0.06704805904155364","0.004935882592690177","-0.004161305550951511","-0.003949102 "1L2J ETC600","0.09012984097353183","-0.01233636905089952","-0.06828176585258916","-0.01509068469749 "2I0G I0G1","0.017258612366276793","-0.001573896166519262","0.018046159180812538","0.007235390599817 "1QKM GEN600","0.06880561851721723","-0.021663096427801065","0.014909053323208354","0.00948922598036 "1U3Q 272501","0.03297848604415776","-0.022831991052953526","-0.029928279720479622","-0.030791792451 "1ERE H2O EST600","1;19;19","1;20;20","1;21;21","1;33;33","1;34;34","1;35;35","1;36;36","1;37;37","1 "1L2J ETC600","2;19;17","2;20;18","2;21;19","2;33;30","2;34;31","2;35;32","2;36;33","2;37;34","2;38; 2I0G I0G1","5;19;17","5;20;18","5;21;19","5;33;30","5;34;31","5;35;32","5;36;33","5;37;34","5;38;35" "1QKM GEN600","3;19;17","3;20;18","3;21;19","3;33;30","3;34;31","3;35;32","3;36;33","3;37;34","3;38; "1U3Q 272501","4;19;17","4;20;18","4;21;19","4;33;30","4;34;31","4;35;32","4;36;33","4;37;34","4;38;
図
12 Save CSV file (Filtered data)の出力(上:エネルギー値、下:インデックス)
ユーザーはエネルギー値に対するファイル名を指定(例:
IFIE_MP2_filtered.csv)し、
それに対応するファイル名(例:IFIE_MP2_filtered_fragment_number.csv)にて、イ
ンデックス情報が出力される(図 12)。出力されるインデック情報は、ポップアップの
情報(図 2)と同一である。
1.6. インストーラ作成
改変した
BioStation Viewer のインストーラを作成した(実施項目(5))。インストーラ
のファイル名は、
BioStationViewerOpen1.0Rev.10.4-setup.exe である。
1.7. 説明書作成
改変した
BioStation Viewer の説明書として本文書を作成した(実施項目(6))。
6
2. ソフトウェア仕様
BioStation Viewer Open1.0 rev.10.4 のソフトウェア仕様について以下に記載する。
2.1. 動作環境
BioStation Viewer Open1.0 rev.10.4 の動作環境を表 1 に示す。ベースとなる
BioStation Viewer 16.02 と同等の動作環境となっている。
表
1 動作環境
項目
要件
OS
Windows 7
CPU
Pentium Ⅳ 2.0GHz 相当以上
メモリ
1GB 以上
HDD
空き容量
100MB 以上
グラフィックボード
VRAM 256MB 以上
2.2. インストール
インストーラをダブルクリックすることでインストールが開始される。インストーラ
の案内に従って進めることで、インストールが完了する。
2.3. 起動
デスクトップに作成されるショートカットアイコン(図 13BioStationViewerOpen1.0)
をダブルクリックすることで、プログラムが起動する。
図
13 ショートカットアイコン
2.4. 操作
追加機能以外の操作方法については、ベースとなる
BioStation Viewer 16.02 と同等
である。インストーラによってインストールされる「BioStationViewer ver. 16.0 ユー
ザーマニュアル」を参照されたい。ユーザーマニュアルへは、プログラムの「Help」メ
ニューからアクセスできる。
7
3. 使用方法
5 種の PDB 構造(PDBID: IERE, 1L2J, 1QKM, 1U3Q, 2I0G)からなるデータを例と
し、アミノ酸配列アラインメント用いた
VISCANA のためのデータ準備と使用方法につ
いて述べる。
なお、
VISCANA の基本的な機能と使用方法については、
「
BioStationViewer ver. 16.0
ユーザーマニュアル」
p.58~p.60 を参照されたい。
3.1. CPF ファイルの準備
FMO 計算を実行して、実行結果の CPF ファイルを取得する。すべての CPF ファイ
ルは同一のフォルダ(以下、データフォルダ)に格納する。
図
14 データフォルダへの CPF ファイルの格納(名前で昇順ソート)
3.2. 配列データの取得
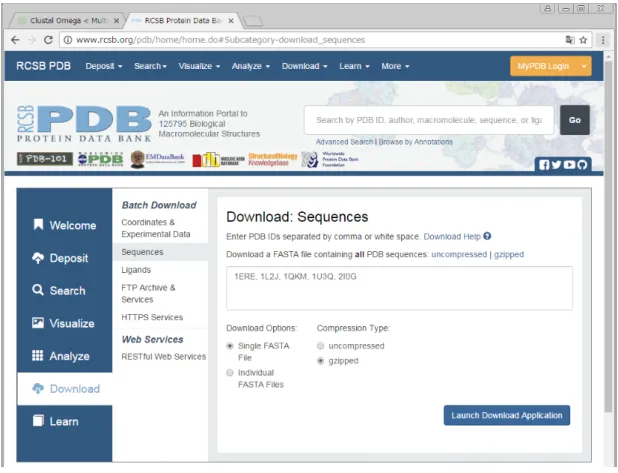
RCSB Protein Data Bank にて、PDBID をもとにアミノ酸配列データを取得する。
PDBID の入力順は、対応する CPF ファイルを Windows のフォルダにて名前で昇順ソ
ートした場合の順序と一致させる。
CPF ファイルの配列データ取得ウェブ画面(図 15)と、取得した配列データ(表 2)
を示す。
8
図
15 Protein Data Bank からデータ取得
表
2 Protein Data Bank からダウンロードした配列データ
>1ERE:A|PDBID|CHAIN|SEQUENCE SKKNSLALSLTADQMVSALLDAEPPILYSEYDPTRPFSEASMMGLLTNLADRELVHMINWAKRVPGFVDLTLHDQVHLLE CAWLEILMIGLVWRSMEHPGKLLFAPNLLLDRNQGKCVEGMVEIFDMLLATSSRFRMMNLQGEEFVCLKSIILLNSGVYT FLSSTLKSLEEKDHIHRVLDKITDTLIHLMAKAGLTLQQQHQRLAQLLLILSHIRHMSNKGMEHLYSMKCKNVVPLYDLL LEMLDAHRLHAPT >1L2J:A|PDBID|CHAIN|SEQUENCE MGSSHHHHHHSSGLVPRGSHMRELLLDALSPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAK KIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLLATTSRFRELKLQH KEYLCVKAMILLNSSMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGME HLLNMKCKNVVPVYDLLLEMLNAHVLRGCKS >1QKM:A|PDBID|CHAIN|SEQUENCE VRELLLDALSPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESC WMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLV TATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEM LNAHVLRGCKSSITG >1U3Q:A|PDBID|CHAIN|SEQUENCE DALSPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLM MGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA DSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNAHVL
9
>2I0G:A|PDBID|CHAIN|SEQUENCE MHHHHHHRELLLDALSPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQV RLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNS SMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVY DLLLEMLNAHVLRGCKS3.3. 配列データの編集
3 次元構造データである PDB ファイルに含まれるアミノ酸残基は、通常、Protein Data
Bank からダウンロードして得られる配列データ(表 2)の一部である。そこで、ダウン
ロードした配列データから
PDB ファイルに含まれる残基のみを切り出したデータを作
成する(表
3)。
配列の
1 行の文字数は、ダウンロードデータ(表 2)と同一でなくても良い。表 3 で
は、各配列を
1 行にまとめている。
表
3 PDB ファイルに含まれる残基のみを切り出した配列データ
>1ERE:A|PDBID|CHAIN|SEQUENCE SLTADQMVSALLDAEPPILYSEYDPTRPFSEASMMGLLTNLADRELVHMINWAKRVPGFVDLTLHDQVHLLECAWLEILMIGLVWRSMEHPGKLLFAPN LLLDRNQGKCVEGMVEIFDMLLATSSRFRMMNLQGEEFVCLKSIILLNSGVYTFLSSTLKSLEEKDHIHRVLDKITDTLIHLMAKAGLTLQQQHQRLAQ LLLILSHIRHMSNKGMEHLYSMKCKNVVPLYDLLLEML >1L2J:A|PDBID|CHAIN|SEQUENCE SPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVL DRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLML LSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA >1QKM:A|PDBID|CHAIN|SEQUENCE SPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVL DRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLML LSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA >1U3Q:A|PDBID|CHAIN|SEQUENCE SPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVL DRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLML LSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA >2I0G:A|PDBID|CHAIN|SEQUENCE SPEQLVLTLLEAEPPHVLISRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVL DRDEGKCVEGILEIFDMLLATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDADSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLML LSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA3.4. アラインメントデータの取得
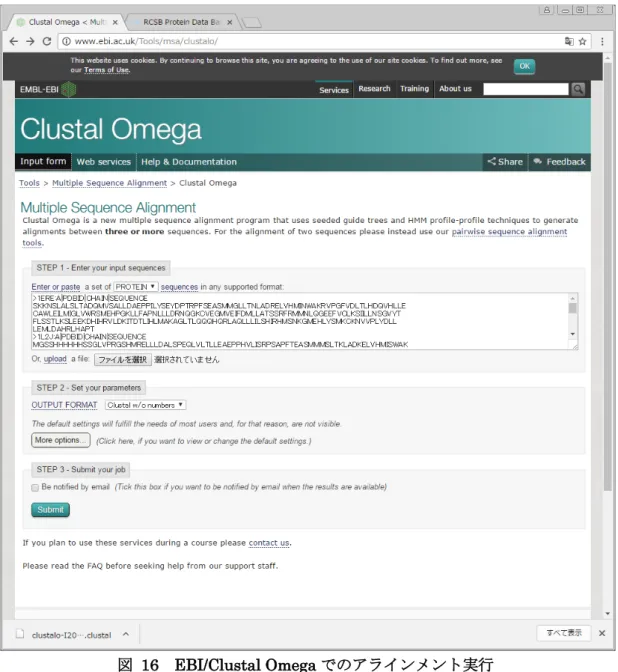
EBI Clustal Omega にて切り出した配列データのアラインメントを行なう。アライン
10
図
16 EBI/Clustal Omega でのアラインメント実行
表
4 アラインメント実行結果
(
clustalo-I20170117-043407-0256-70767858-pg.clustal)
CLUSTAL O(1.2.4) multiple sequence alignment
1ERE:A|PDBID|CHAIN|SEQUENCE SLTADQMVSALLDAEPPILYSEYDPTRPFSEASMMGLLTNLADRELVHMINWAKRVPGFV 1L2J:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 1QKM:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 1U3Q:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 2I0G:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV : :*:* :**:**** : *: **:***** **:***:******.***::**** 1ERE:A|PDBID|CHAIN|SEQUENCE DLTLHDQVHLLECAWLEILMIGLVWRSMEHPGKLLFAPNLLLDRNQGKCVEGMVEIFDML 1L2J:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML 1QKM:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML 1U3Q:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML
11
2I0G:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML :*:*.***:***..*:*:**:**:***::*****:***:*:***::******::****** 1ERE:A|PDBID|CHAIN|SEQUENCE LATSSRFRMMNLQGEEFVCLKSIILLNSGVYTFLSSTLKSLEEKDHIHRVLDKITDTLIH 1L2J:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 1QKM:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 1U3Q:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 2I0G:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW ***:**** ::** :*::*:*::*****.:* ::::* .: :.. :: ::*: :**:*: 1ERE:A|PDBID|CHAIN|SEQUENCE LMAKAGLTLQQQHQRLAQLLLILSHIRHMSNKGMEHLYSMKCKNVVPLYDLLLEML-- 1L2J:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA 1QKM:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA 1U3Q:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA 2I0G:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA ::**:*:: *** ***:**::***:** ******** .********:********3.5. アラインメントデータの編集
アラインメントデータにて、リガンドや水分子などアミノ酸配列以外の分子のフラグ
メント位置情報を記述する。
FMO 計算に用いた PDB ファイルの全てにおいて、リガンドなどの注目するフラグメ
ントがアミノ酸フラグメントの直後にある場合には、アラインメントデータファイル
(
*.clustal、表 4)を直接 VISCANA の入力として用いることができる(本節の以下の
手順は不要)
。
上記以外の場合には、リガンドなどの注目するフラグメントの位置を合わせる必要が
ある。位置合わせには、表
5 の末尾の
赤字の様に配列文字を追加して調整する。配列文
字の追加ルールは以下の通り。
i.
全てのアラインメント配列文字の数を同一とする。
ii.
フラグメントが無い場合には、ギャップ文字(
-)を記述する。
iii.
フラグメントがある位置には、ギャップ文字以外の任意文字を記載可能である。
※
記録目的のため、通常のアミノ酸文字列を避け、任意のアミノ酸を表す配列文
字(
X)やリガンドなどの注目するフラグメント位置を特別な文字(@,#,$,…)で
表しておくとよい。
iv.
リガンドなどの注目するフラグメント位置(複数ある場合は最後のフラグメント)
の直前までの記載でよい。
v.
一行の配列文字数が定型(
30 文字)を超えてもよい。
12
表
5 アラインメントデータの編集
(
00_clustalo-I20170117-043407-0256-70767858-pg.clustal)
CLUSTAL O(1.2.4) multiple sequence alignment
1ERE:A|PDBID|CHAIN|SEQUENCE SLTADQMVSALLDAEPPILYSEYDPTRPFSEASMMGLLTNLADRELVHMINWAKRVPGFV 1L2J:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 1QKM:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 1U3Q:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV 2I0G:A|PDBID|CHAIN|SEQUENCE --SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFV : :*:* :**:**** : *: **:***** **:***:******.***::**** 1ERE:A|PDBID|CHAIN|SEQUENCE DLTLHDQVHLLECAWLEILMIGLVWRSMEHPGKLLFAPNLLLDRNQGKCVEGMVEIFDML 1L2J:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML 1QKM:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML 1U3Q:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML 2I0G:A|PDBID|CHAIN|SEQUENCE ELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDML :*:*.***:***..*:*:**:**:***::*****:***:*:***::******::****** 1ERE:A|PDBID|CHAIN|SEQUENCE LATSSRFRMMNLQGEEFVCLKSIILLNSGVYTFLSSTLKSLEEKDHIHRVLDKITDTLIH 1L2J:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 1QKM:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 1U3Q:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW 2I0G:A|PDBID|CHAIN|SEQUENCE LATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVW ***:**** ::** :*::*:*::*****.:* ::::* .: :.. :: ::*: :**:*: 1ERE:A|PDBID|CHAIN|SEQUENCE LMAKAGLTLQQQHQRLAQLLLILSHIRHMSNKGMEHLYSMKCKNVVPLYDLLLEML--X@ 1L2J:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@ 1QKM:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@ 1U3Q:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@ 2I0G:A|PDBID|CHAIN|SEQUENCE VIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@ ::**:*:: *** ***:**::***:** ******** .********:********
3.6. アラインメントデータの配置
データフォルダにアラインメントデータ(
*.clustal)を格納する(図 17)。アライン
メントデータが複数ある場合には、名前・昇順ソートで最初のデータが使用される。
図
17 では編集を行なったアラインメントデータについて、そのファイル名の先頭を
「
00_」とし、VISCANA 実行時に読み込まれるようにしている。
13
図
17 データフォルダへのアラインメントデータの格納
3.7. VISCANA 実行
BioStation Viewer の「Monitor」メニューから「VISCANA」を選択して VISCANA
ウィンドウを開く(図
18)。
VISCANA のウィンドウにて、
「
CPF Data Directory…」ボタンをクリックしてデータ
フォルダを設定する。次いで「
Load」ボタンをクリックすると、データフォルダのアラ
インメントデータおよび
CPF が読み込まれる。
「
Apply」ボタンをクリックするとクラスター解析が行われ、結果が表示される(図
19)。
14
図
19 VISCANA ウィンドウ(アラインメント実行結果)
左下の「
Data Select」から「IFIE」を選び、プルダウンから「MP2」を選び、
「
Apply」
ボタンを押すと図
20 のようになる。
図
20 VISCANA ウィンドウ(IFIE, MP2 データ選択実行)
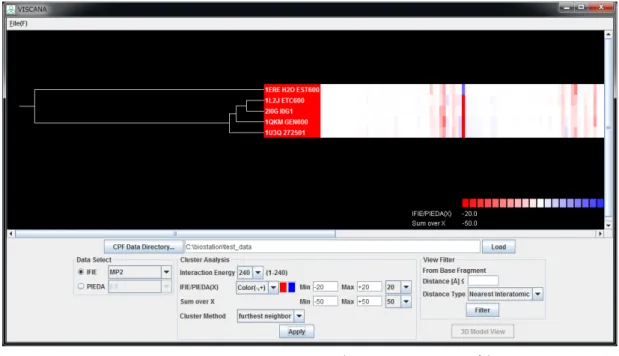
表示される色のスケールを変更するには、中央下のエネルギーの下限「
Min」・上限
「
Max」を設定する。一度「Apply」ボタンを押すとデータの下限・上限が表示される。
15
を
-20、上限を+20 に設定、
「
Sum over X」の右側のプルダウンから 50 を選び下限を-50、
上限を
50 に設定し、「Apply」ボタンを押すと、図 21 のように表示される。
図
21 VISCANA ウィンドウ(カラースケール設定)
Interaction Energy の起点となるフラグメントからの距離により、表示するフラグメ
ント数を絞るためには、右下の「
View Filter」の「Distance」に値を設定する。
「
Distance」
に
15(Å)を設定して、「Filter」ボタンを押すと、図 22 のようにフラグメントが限定
されて表示される。
16
VISCANA 実行後には、アラインメント配列情報を記載したファイル(sequence.txt)
が出力される(図
23)。sequence.txt には、アラインメントデータファイルの名前、配
列の本数、各配列の
PDBID・Chain、配列文字列、アミノ酸残基数、配列文字数が出力
される(表
6)。
図
23 データフォルダ(VISCANA 実行後)
表
6 sequence.txt
C:\test_data\00_clustalo-I20170117-043407-0256-70767858-pg.clustal #sequences : 51ERE|A|SLTADQMVSALLDAEPPILYSEYDPTRPFSEASMMGLLTNLADRELVHMINWAKRVPGFVDLTLHDQVHLLECAWLEILMIGLVWRSMEHPGK LLFAPNLLLDRNQGKCVEGMVEIFDMLLATSSRFRMMNLQGEEFVCLKSIILLNSGVYTFLSSTLKSLEEKDHIHRVLDKITDTLIHLMAKAGLTLQQQ HQRLAQLLLILSHIRHMSNKGMEHLYSMKCKNVVPLYDLLLEML--X@(#=238/240) 1L2J|A|--SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLL ATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@(#=235/240) 1QKM|A|--SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLL ATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@(#=235/240)
1U3Q|A|--SPEQLVLTLLEAEPPHVLI
-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLL ATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@(#=235/240) 2I0G|A|--SPEQLVLTLLEAEPPHVLI-SRPSAPFTEASMMMSLTKLADKELVHMISWAKKIPGFVELSLFDQVRLLESCWMEVLMMGLMWRSIDHPGKLIFAPDLVLDRDEGKCVEGILEIFDMLL ATTSRFRELKLQHKEYLCVKAMILLNSSMYPLVTATQDA-DSSRKLAHLLNAVTDALVWVIAKSGISSQQQSMRLANLLMLLSHVRHASNKGMEHLLNMKCKNVVPVYDLLLEMLNA-@(#=235/240)