STR 開発講習会資料
文責:山 曜(京都大学大学院理学研究科) 初版:2014/5/30 改訂版:2016/2/4
資料内容
作業内容
A.VirtualBox と Linux の環境設定 ・・・・・・・・・・・・・・・・・p.1
B.QDD3 を用いた NGS データからの STR 解析用プライマー設計・・・p.5
C.優良プライマーの選別と Multiplex 系の構築・・・・・・・・・・・・p.11
はじめに
この資料の作成にあたり,武島弘彦博士,武藤望生博士,STR 開発セミナー参
加者の方々に大変お世話になりました.この場を借りてお礼申し上げます.
作業内容
A.VirtualBox と Linux の環境設定
VirtualBox は,ホストの OS(Win,mac など)上でゲストの OS(Linux など)を動 かすためのソフトです.今回の STR 開発ではソフトウェア QDD(Meglécz et al., 2010, 2014)を使用して STR のプライマーを設計します.すでに QDD がインストールされ ている LinuxOS が配布されていますので,今回はそれを VirtualBox で動かして解析し ます.QDD で解析する前にまずは,Linux 上のネットワーク環境やホストとゲストの OS 間での共有フォルダの設定などを行って解析の下準備を行います.この過程は一度 設定すれば,繰り返し行う必要はありません.
1.
VirtualBox のインストールと Linux の読み込み
参考 URL 全般 http://net.imbe.fr/ emeglecz/qdd_installation.html VirtualBox のインストール: http://www.kkaneko.com/rinkou/virtualbox/virtualbox.html ① VirutalBox の Web ページからダウンロードした実行ファイル 「VirtualBox-4.3.12-93733-win.exe」をクリックします.ウィザードに従ってイ ンストールします.設定はデフォルトで OK です.② QDD3 の Web ページから QDD-VM version3.1.1 をダウンロードします.容量が 重いので時間がかかります.ダウンロードしたファイルはパソコンの適当な場所に 置いてください.この QDD-Galaxy 3.1.1 は LinuxOS の一種である ubuntu に, QDD などの解析ソフトがすでにインストールされている状態のものです. ③ インストールした VirutalBox を起動します.Windows7 で起動しない場合,右ク リックで VirtualBox のプロパティを開き,互換モードでいずれかの OS を選択する とよい場合があります.起動後,「ファイル→仮想アプライアンスのインポート」を 選択します.設定はデフォルトで大丈夫です.仮想アプライアンスには,「QDD-VM」 を選択してください.成功すれば,VirtualBox マネージャー上に
「QDD-Galaxy-3.1.1」が現れます.windows にて,ubuntu が 32bit として読み込 まれてしまい,起動できなくなる問題が確認されています.執筆者は mac なので詳 しくはわからないのですが,bios の設定を変更すると 64bit として読み込めるよう になるそうです.詳しくはググってください. ④ 「設定」をクリックし,「システム」タブから自分のパソコンのスペックに見合った CPU(「マザーボード」タブから)やメモリ(「プロセッサー」タブから)を割り当 ててください.メモリや CPU を多く割り当てると,それだけ解析時間が早くなり ます.念のため,デフォルト以下の値にはしないでください.
2.
Linux 上での各種設定
① 画面左のアイコンをダブルクリックして QDD-Galaxy を起動してください.パスワ ードは qddGalaxy です.デ フ ォ ル ト で は キ ー ボ ー ド の 配 列 が フ ラ ン ス 語 に な っ て い ま す の で , 右 上 の ア イ コ ン を ク リ ッ ク し 英 語 を 選 択 し て か ら 入 力 し て く だ さ い . ② 日本版のキーボード配置に設定し直します.左側の「System Setting→Keyboard Layout」を選択してください.左下の「+」マークをクリックして,表示されたリ ストから日本版のキーボード配置を選択し,「add」をクリックして使用リストに加 えください.フランス語や英語のキーボード配置は「」を押して削除するほうが よいと思います.不要な配置を削除しない場合,デスクトップの右上のキーボード のアイコンから,Japanese を選択する必要があります.③ Linux 上での操作性を改善するため,Gest Additions をインストールします.ホス ト側のタブから「Device→Insert Gest Additions CD image」(日本語表示の場合「デ バイス→「Guest additions の CD イメージを挿入」)を選択します.Linux 側から
のメッセージに従ってインストールを実行してください.パスワードは qddGalaxy です.
④ オプション
ネットワークを設定します.左のアイコンから「System Setting→Network→ Network proxy→Automatic」を選択し, http://wpad.kuins.net/proxy.pac と記 入し Apply system wide をクリックして適用してください(Fig.1).このアドレス 部分は京大内での設定になります.他の機関の内部で使用される場合,その機関内 でのプロキシ設定方法に準じてください(自動プロキシ設定についての項目があり ましたら,そこに記載されているアドレスを入力すれば大丈夫と思います). ⑤ FireFox を立ち上げて Web ページが開けることを確認してください.
3. ホスト OS と Linux 間の共有ファイルの設定
① ホストと Linux 間でデータ交換をするためのフォルダをホスト側に設置します.ホ ス ト の 適 当 な 位 置 に 新 規 の フ ォ ル ダ を 設 置 し て く だ さ い . こ の 資 料 で は , 「qddGalaxy」と名付けたとして話を進めます. ② VirtualBox マネージャーの「設定→共有フォルダ」を選択し,ウィンドウ右端の小 さいフォルダアイコン「新規共有フォルダを追加する」を選択します.さきほど作 ったフォルダを選択し,「永続化する」にだけチェックを入れます. Fig. 1 上記のように入力してください.③ Linux 側にも同じ名前のファイルを作成します.特にこだわりが無ければ,home に作成されることをお薦めします.
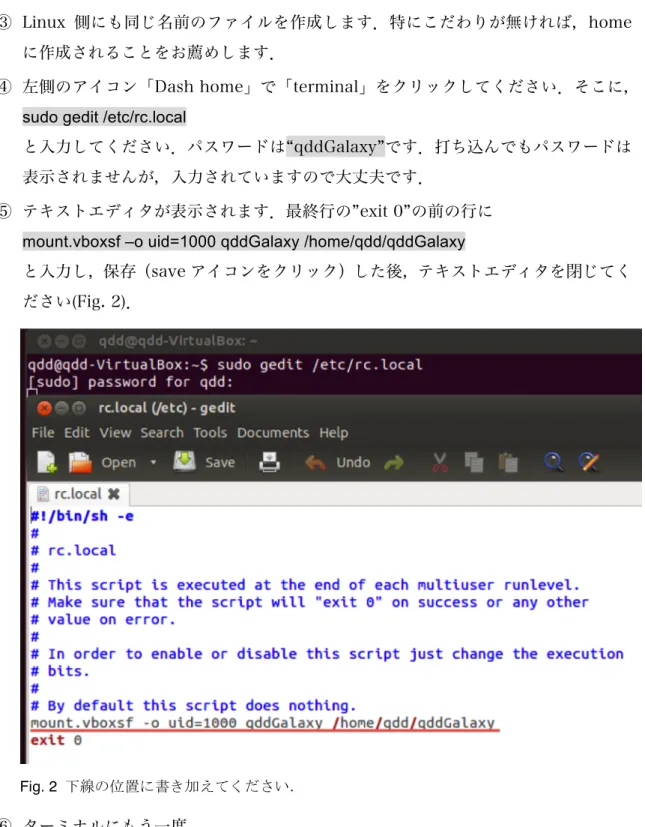
④ 左側のアイコン「Dash home」で「terminal」をクリックしてください.そこに, sudo gedit /etc/rc.local
と入力してください.パスワードは qddGalaxy です.打ち込んでもパスワードは 表示されませんが,入力されていますので大丈夫です.
⑤ テキストエディタが表示されます.最終行の exit 0 の前の行に mount.vboxsf –o uid=1000 qddGalaxy /home/qdd/qddGalaxy
と入力し,保存(save アイコンをクリック)した後,テキストエディタを閉じてく ださい(Fig. 2).
⑥ ターミナルにもう一度,
mount.vboxsf –o uid=1000 qddGalaxy /home/qdd/qddGalaxy
と入力してください.ホスト側からフォルダ「qddGalaxy」に解析する FASTA フ ァイルを移動してみて,Linux 側からもそのファイルが開けることを確認してくだ さい.
B. QDD3 を用いた NGS データからの STR 解析用プライマー設計
STR 領域のプライマーを設計するソフトは幾つかあるようですが,今回は QDD3 と いうソフトを使用します.QDD3 は Perl 言語で書かれた以下の 4 つの解析プログラム から構成されています. 1. STR 領域の抽出 2. コンセンサス配列の作成 3. プライマーの作成 4. BLAST 今回は 3 までを実施します. QDD による解析を一度中断した場合(ウェブブラウザを閉じた場合)は,この項目を 始めから実行してください. 参考 URL 全般 http://net.imbe.fr/ emeglecz/qdd_run.html Galaxy について http://www.nabe-intl.co.jp/takeruwiki/index.php?Galaxy を動かしてみました http://blog.amelieff.jp/?day=20120807① Galaxy サーバーを起動します.Galaxy はゲノムデータを,web ブラウザを利用し GUI で動かすためのシステムです.Galaxy 内には多数のソフトが内包されていま す.QDD の web ページからダウンロードした ubuntu には QDD がインストール されている Galaxy サーバーを搭載しているため,容易に解析が行えます.まずは Dash home から terminal を起動し,
cd ~/galaxy-dist/ と入力してください.次に sudo sh run.sh と入力してください.パスワードが要求されますので,また qddGalaxy と入力し てください. ② terminal に「serving on http//127.0.1:8080」と表示されるまで待ってください. しばらく時間がかかります.
③ 表示されましたら,FireFox を起動します.URL 入力欄に http://127.0.0.1:8080/ と入力してください.FireFox に Galaxy サーバーが表示されます (Fig. 3).
④ アカウントに入ります.ウィンドウ上部の User から入ります. Email: qddGalaxy@gmail.com Pass:qddGalaxy です.アカウントに入ることで,解析の保存ができるようになります.解析結果は 要所要所で自動的に保存されます.保存内容は User→saved history から閲覧でき ます. ⑤ 新規の解析セッションを作成します.右上の歯車マークをクリックし,「Create New」を選択してください.新規に名前を付けてください.
⑥ データを読み込ませます.左端の Tools 欄の上から 2 番目に「Get Data」がありま
す.その中の「Upload File」を選択してください.「Browse」をクリックし,画面
右の「Places」から「qdd」を選び,ホストとの共有フォルダ「qddGalaxy」中に 置いてある,解析に用いる FASTA ファイルを選択します.最後に「Execute」を クリックすると,ファイルが Galaxy サーバーに upload されます.NGS からはき 出された file を使用する場合,予めタグ配列を除去し,Galaxy に upload してから Fig. 3 terminal に上記の表示が出たら,FireFox に http://127.0.0.1:8080 と入力してください.
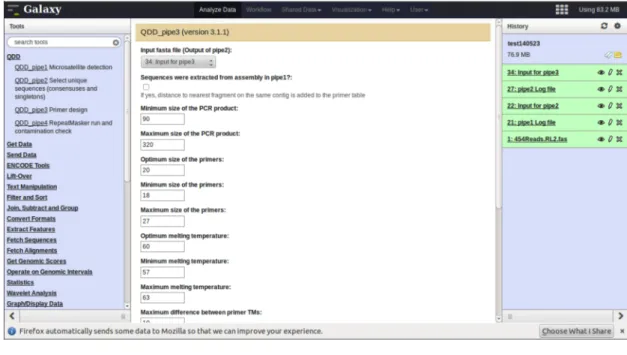
FASTA 形式に変換してください.sff や fastq 形式から fasta 形式への変更は Galaxy 内で行えます(Convert Formats から行えます). pipe ごとに解析を行う ⑦ 解析を開始します.まずは STR 領域の探索です.QDD の pipe1では,input ファ イルの中から STR 領域を探索し,また一定以下の長さの配列を除外する解析を行い ます.「Tools」の一番上にある 「QDD」をクリックしてください.その中の 「QDD_pipe1」をクリックしてください.表示された画面上で,以下の様に選択し てください.
Input fasta file: 解析する FASTA ファイル
Input sequence are already assembled: yes(未アセンブルデータの場合は no) Flanking region length: 200~300
Sequence length limit: 80
選択が終わったら,「Execute」をクリックしてください.解析が終了すると,右端
の「history」に新しくファイルが表示され,黄色から緑色になります(Fig. 4).灰 色の場合は histrory 右の更新ボタンをクリックします.
⑧ 次はコンセンサス配列の作成ですが,今回使用するデータはすでにアセンブルされ たデータです.「Tools」から「QDD_pipe2」を選択してください.「Sequence with Fig. 4 pipe1 解析画面の様子
microsatellites」が「Input for pipe2」になっていることを確認し,Make consensus sequences? は No を選択し,「Execute」をクリックしてください.もし NGS デ ータをそのまま解析する場合,配列データの中には同じ領域が複数本含まれる場合 があります.それらを把握して,重複した配列を無くす必要があります.そのとき は Make consensus sequences? を yes にしてください.解析終了まで少し時間 が掛かります.「History」のファイルがグレーのときは,上の更新ボタンを押して ください. ⑨ 最後はプライマーの設計です.pipe3 は,設定されたパラメータに基づいてプライ マーを設計します.プライマー長やアニーリング温度などを入力します.入力し終 わったら,また「Execute」をクリックします.解析には時間が掛かります(デー タ量次第ですが,今回のミツバチゲノムデータの場合数時間くらい)(Fig. 5).ユニ バーサル蛍光プライマーのアニーリング温度が 59℃ですので,それを大きく異なる 温度設定にはしないでください.
⑩ 解析が終了したら,ファイル「Table with primer」をクリックし,次いでフロッピ ーマークをクリックしてこのファイルをダウンロードしてください.ホーム内のダ ウンロードフォルダに保存されます.ダウンロードされたファイルをホスト側に移 動させ,エクセルで開きます.各行が設計されたプライマーと,それに関する情報 を表しています.
Workflow 機能を利用した解析
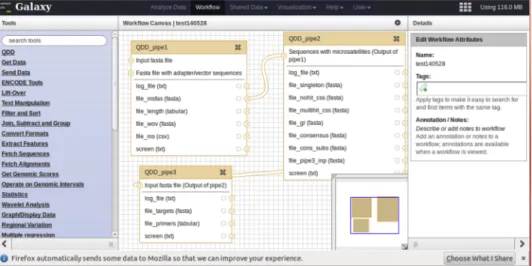
上記では pipe ごとに解析を実行しましたが,下図のような Workflow 機能を使えば, 一度に全ての変数を設定し,解析を実行することができるため非常に楽です. (Fig. 6).
⑪ Workflow の管理画面に移ります.ブラウザの上方にある Workflow をクリッ
クします.
⑫ Create new workflow をクリックします.workflow name を記入し,create
をクリックします.すると管理画面に作成した workflow が追加されます.
⑬ workflow の内容作成画面に移ります.作成した workflow に表示されている
下向き三角をクリックし,edit を選択します.
⑭ workflow を作成します.左端の Tools に,Galaxy に含まれるプログラム群
が表示されています.workflow 機能ではこれらの解析を予め繋ぎ,変数を設
定します.このことで,スタートボタンを押すだけで解析が完了します.ま
ず Tools 内の QDD をクリックし,続いて QDD_pipe1 をクリックします.画
面中央には QDD_pipe1 の input file と out file の内容が表示されます.右端に
は,QDD_pipe1 で設定する変数とその変数の説明が表示されます.”Input
sequences are already assembled”は,今回はすでにアセンブルされているデ
ータを用いますので,yes を選択してください.
⑮ QDD_pipe2 をクリックします.すると,同じように QDD_pipe2 が表示され
ます.QDD_pipe1 の out putfile のうち,”file_msfas”を pipe2 渡しますの
で,”file_msfas”の右端矢印をつかみ,pipe2 の”Sequences with microsatellites
(Output of pipe1)”の左端の矢印に持って行きます.今回のデータはすでにア
センブルされていますので,”Make consensus sequences?”は No を選択しま
す.
⑯ QDD_pipe3 を ク リ ッ ク し ま す . さ き ほ ど と 同 様 に , QDD_pipe2
の”file_pipe3_inp”の矢印を QDD_pipe3 の”Input fasta file”に接続します.
QDD_pipe3 の変数はプライマー設計に関するものになります.各自設定くだ
さい.
⑯ この workflow を保存します.右上の歯車マークをクリックし,save を選択
します.
⑰ 解析を開始します.同じ歯車マークの中の Run を選択します.次に表示され
た画面で,Input fasta file で解析したいファイルを選択します.最後に Run
workflow をクリックすると解析が開始されます.
C.優良プライマーの選別と Multiplex 系の構築
QDD で作成された候補プライマーは大量です.その中から優良なプライマーを選別 し,Multiplex 系を構築できるような組み合わせを抽出する必要があります.選別の方 法は一つではありませんが,今回はその一つの考え方と実際の手順を説明します.他に も QDD の HP にある How to choose primers from the primer table. も参考にして ください. ① STR 領域が単純なものを選別します.QDD は領域の複雑さごとに,A∼G までのラ ンク分けを行います.今回候補は大量にあるはずなので,けちらずに単純で解析し やすい領域だけを抽出します.もっとも単純なのはランク A です(詳しくはマニュ アル p.39 を参照).Excel のフィルター機能で「Design」の列を並べ替えて,A ラ ンクのものだけをコピーし,新しいシートにペーストしてください (Fig. 7).フィ ルター機能で要らない行を隠しても大丈夫です. ② すでに一定以上のリピートが確認されているものを選びます.あまりにリピート数 が 短 い も の は , 多 型 性 が な い 可 能 性 が あ る か ら で す . 「TARGET_MS_LENGTH_IN_REPEAT_NUMBER」を並べ替えて,一定以上のリ ピート数が確認されたプライマーデザインだけをコピーし次のシートにコピーしま しょう.参考に,筆者は 9 回以上のものを選びました. ③ STR 領域に近すぎる位置に設計されたプライマーはあまり使いたくないので,それ らを排除します.「MIN_PRIMER_TARGET_DIST」は STR 領域と設定されたプラ イマー領域の間の距離を表しています.これが一定以上のものだけ選択し,新しい シートに移します.参考に筆者は 6bp 以上離れたものを選択して以降の解析に回し ました. ④ 各 STR 領 域 に つ い て 最 も 優 良 な プ ラ イ マ ー を 選 択 し ま す . 「ONE_PRIMER_FOR_EACH_SEQ」が「1」のプライマーは,計算上ではそのリ ードに対してもっとも上手く設計されたプライマーとなります.この列が1のもの だけを選択し,次のシートにコピーしてください. Fig. 7 シートを増やしながらソートしていきます.
⑤ ここで,マルチプレックス系の構築について一度考えます.4 色の蛍光を使えるの であれば,大体 12 領域程をマルチプレックスできます.100bp 付近,200bp 付近, 300bp 付近にそれぞれ 4 領域,といった具合です.残った候補プライマーを 「PCR_PRODUCT_SIZE」順に並べ,このようなマルチプレックス系が何個組めそ うか考えます.そして必要予定数に到達していれば,そのままマルチプレックスを 実際に組みます.必要予定数のプライマーが残っていなければ,今までのソートの 過程を緩めてもう一度ソートし直すか,ピンポイントで欲しいプライマー(例えば, AC 以外のリピート領域など)を選び出してきます. ⑥ 実際にマルチプレックス系を組みます.多型の範囲を「PCR_PRODUCT_SIZE」の 50bp 以内と見積もり,同じ蛍光を付ける予定のもの同士で「PCR_PRODUCT_SIZE 50bp」が重複しないように設計します.「PCR_PRODUCT_SIZE」の前に新しく 2 つの列を追加し,それぞれ「PCR_PRODUCT_SIZE」に 50 足した値と引いた値を 入力します.この 3 つの列を選択し,Excel の株価グラフを作成します (Fig. 8). このグラフを確認しながら,どのプライマーにどの色をつけるか,また産物範囲が かぶった場合は他の候補と入れ替えながらマルチプレックスの組み合わせを考えて いきます.scaffold 上の領域は連鎖しているため,同じ scaffold からは 1 つの STR 領域のみを選ぶようにしてください. Fig. 8 株価グラフで書いたマルチプレックス設計図.+50bp を高値,-50bp を安値, 0 50 100 150 200 250 300 350 400 450 +50 -50 PCR_PRODUCT_SIZE
product size を終値として指定し,グラフを作成する.
⑦ 各プライマー配列の forward 側にユニバーサル蛍光プライマー用の配列(Blacket et al., 2012)を,reverse 側に PIG-tailing (GTTTCTT)を付加し,マルチプレ ックス可能かどうかを再度確認します.ここでは MultiplexManager (Holleley and Geert, 2009)というソフトを使用し,アニーリング温度,1マルチプレック ス 内 で の プ ラ イ マ ー の 組 み 合 わ せ の 善 し 悪 し な ど を 確 認 し ま す . MultiplexManager は web ページよりダウンロード,ウィザードに従ってインスト ールしてください.MultiplexManager 用のインプットファイルを作成します. MultiplexManager を起動し,表示された項目について新しい Excel シートに作成 していきます. 「Chromosome」は1,「Genetic location」「Heterozygosity」は 0,Number of Alleles は1,「Annealing Temperature」は空白とし,「Forward Sequence」はユニバーサル蛍光プライマーの配列を加えたものとしてください. ⑧ 作成した Excel シートを csv 形式で保存し,MultiplexManager に読み込ませます. 読み込みが成功したら,Utilities の「Calculate All Annealing Temperatures」を 選択し,アニーリング温度を計算します.次に 「Dyes and Option」を選択し,各 プライマーの蛍光を選択します.最後に「Analyse Multiplex」を選択し,解析を開 始します. ⑨ 解析結果について,アニーリング温度が極端に違わないこと,同じ色のプライマー 同士で重複する領域がないこと,プライマー同士が結合しにくいことなどを確認し てください (Fig. 9). 以上で解析は終了になります.あとはプライマーを注文するだけです.お疲れ様でした.
補足
今回のマルチプレックス系の構築方法はかなり大雑把な方法です.各マルチプレック スで 12 個すべてが使えることはあまりないと思いますし,解析を始めてから蛍光が重 複する場合が出てくる可能性もあります.時間をかけてもいいので,より丁寧で解析し Fig. 9 このような結果が出てきます.やすいマルチプレックス系を構築したい場合は Guichoux et al. (2011)や,陶山 (2012) を参考にしてください.
引用文献
Blacket, M.J., Robin, C., Good, R.T., Lee, S.F., Miller, A.D., 2012. Universal primers for fluorescent labeling of PCR fragment–an efficient and cost–effective approach to genotyping by fluorescence. Mol. Ecol. Resour. 12, 456-463.
Guichoux, E., Lagache, L., Wagner, S., Chaumeil, P., Leger, P., Lepais, O., Lepoittevin, C., Malausa, T., Revardel, E., Salin, F., Petit, R.J., 2011. Current trends in microsatellite genotyping Mol. Ecol. Resour. 11, 591-611.
Holleley, C.E., Geerts, P.G., 2009. Multiplex Manager 1.0: acrossplatform computer program that plans and optimizes multiplex PCR. Bio Techniques, 46, 511-517. Kenta, T., Gratten, J., Haigh, N.S., Hinten, G.N., Slate, J., Butlin, R.K., Burke, T., 2008.
Multiplex SNP-SCALE: a cost-effective medium-throughput single nucleotide polymorphism genotyping method. Mol Ecol. Resour. 8, 1230-1238.
Meglécz, E. Costedoat, C., Dubut, V., Gilles, A., Malausa, T., Pech, N. and Martin J-F. 2010. QDD: a user-friendly program to select microsatellite markers and
designprimers from large sequencing projects. Bioinformatics, 26(3) 403–404. Meglécz, E., Pech, N., Gilles, A., Dubut, V., Hingamp, P., Trilles, A., Grenier, R. and
Martin, JF. 2014. QDD version 3.1: A user friendly computer program for microsatellite selection and primer design revisited: experimental validation of variables determining genotyping success rate. Mol. Ecol. Resour. doi: 10.1111/1755-0998.12271.
陶山佳久, 2012. SSR ジェノタイピング手法. in: 津村義彦,陶山佳久(編)森の分子 生態学2,文一総合出版,東京,pp.291-323