Title
腸内に生息する細菌の系統( 本文(Fulltext) )
Author(s)
江崎, 孝行
Citation
[腸内細菌学雑誌 = Journal of intestinal microbiology] vol.[20]
no.[3] p.[237]-[244]
Issue Date
2006-07-01
Rights
The Japan Bifidus Foundation (日本ビフィズス菌センター)
Version
出版社版 (publisher version) postprint
URL
http://hdl.handle.net/20.500.12099/29992
細菌は 1980 年代の中頃から蓄積されてきた 16S rDNA
の配列に基いて再分類され,分類体系が大幅に変化した
(26, 27)
.記載された細菌種は現在で約 5500 種に達する
が,その 3 割近い菌種が,再分類の対象となった.
その結果,16SrDNA の系統分類では原核生物は古細
菌 Archaea と真正細菌 Eubacteria(or Bacteria)に分
かれた.配列だけの登録ではなく生きた菌が記録されて
いる古細菌 Archaea は Euryarchaeota と Crenarchaeota
の 2 つの門 Phylum に,真正細菌 Eubacteria は 24 の門に
分類されている.ヒトの口腔から大腸までの消化管に生
息 す る 菌 種 は ,こ の 両 方 の domain に 分 布 し て い る
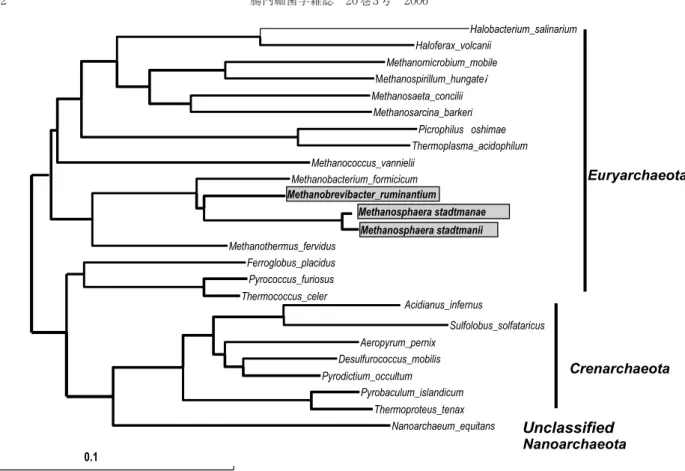
(Fig. 1,Archaea 系統樹)
.
古細菌 Domain Archaea
湖沼の汚泥,海底火山,高温の温泉などに生息する
Archaea
の系統に分類される原核生物がヒトの腸管にも
生息するというデータは早くから蓄積されてきた(4,
14
, 18, 19)
.メ タ ン 生 産 菌 と し て 大 腸 に 生 息 す る
Archaea
は硫酸還元菌である真正細菌の Desulfovibrio
とリンクさせて大腸発癌研究の標的となってきた(16)
.
しかし,多くのデータがメタンの生産を確認しているだ
けで菌種名を同定した論文は少ない.これは Archaea
の培養が困難であるがメタンの測定が容易であることに
起 因 す る .腸 管 に 生 息 す る メ タ ン 生 産 菌 と し て は
Methanobrevibacter smithii
(Table 2)が最も多くの記
載があり(18)
,発癌物質の蓄積と関連づけた研究が多
い (11)
.そ の ほ か の 菌 種 と し て は Methanosphaera
stadtmaniae
(14)が 報 告 さ れ て い る .い ず れ も
Archaea
の Euryarchaea 門に分類される.
培養が困難なメタン生産菌の分子系統が整備され,
PCR 法や Fish 法による解析(1, 3)が容易になったこと
から,菌種の記載は少ないが,腸管のフローラ解析のデ
ータが多く発表されている.特に Raskin ら(17)が 8
種類のメタン生産菌のプローブを報告し,解析に拍車が
かかったので,今後は菌種レベルの報告が蓄積されてく
ればより詳細なメタン生産菌の腸管内の分布が解明され
ると期待される.
真正細菌(Domain Eubacteria あるいは Bacteria)
Domain Eubacteria は新しく発表される Bergy’s
man-ualでは 24 の門 Phylum(Table 1)に分類される予定に
なっている.そのうち消化管内フローラを形成する菌種
は Firmicutes,Actinobacteria,Fusobacteria,
Fibrobacteres
,Proteobacteria,Treponema お よ び
Bacteroidetes
の 7 つの門 Phylum に分布する.グラム陰
性菌では Bacteroidetes 門の菌群が優位を占め,そのな
か で Genus Bacteroides の 菌 種 で あ る B. vulgatus,
B. distasonis
,および B. eggerthii が大腸内フローラを
構成する系統としては優位な菌である(Table 2a)
.一
方,かって Bactroides 属に分類されていた Prevotella 属,
および Porphylomonas 属の菌群は主に口腔内フローラ
を形成している.グラム陰性の Fusobacterium 属の菌種
に分類されていた F. prausnitzii は大腸内優位フローラ
の形成菌種として報告されていたが,Faecalibacterium
prausnitzii
としてグラム陽性の Firmicutes 門に再分類
されたため,Fusobacterium 属の菌種が大腸内フロー
ラ の 優 位 菌 と し て 報 告 さ れ る こ と は 少 な く な っ た .
Fusobacterium
属では F. nucleatum が口腔内フローラ
を形成している.
大腸内フローラの優位菌であった Bacteroides
suc-腸内に生息する細菌の系統
江崎 孝行
* *岐阜大学大学院医科学系病原体制御分野
Phylogenetic Position of Human Intestinal Bacteria
Takayuki EZAKI
*
*Department of Microbiology, Gifu University Graduate School of Medicine, Yanagido 1–1, Gifu 501–1194, Japan
腸内細菌学雑誌
20 : 237–244,2006
2005 年 12 月 7 日受付
*〒 501–1194 岐阜市柳戸 1–1
1–1 Yanagito, Gifu 501–1194,
Japan
実験講座
cinogenes
も Fibrobacteres 門 に 再 分 類 さ れ ,
Fibrobacter succinigenes
として独立した系統として位
置ずけられている(Fig. 2 グラム陰性菌の系統樹)
.
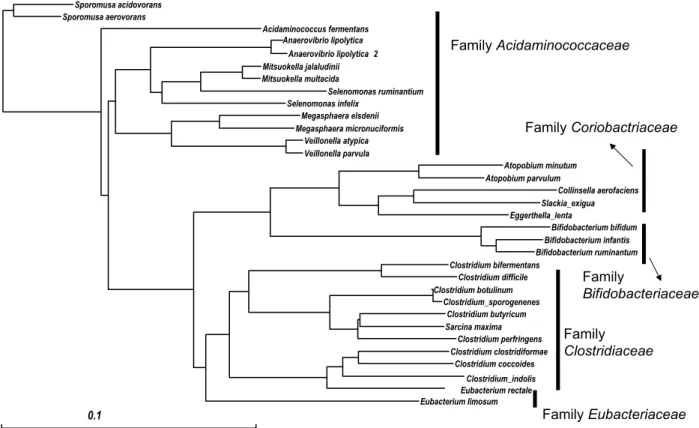
グラム陽性菌は DNA の GC 含量が高い Actinobacteria
門 と GC 含 量 が 低 い Firmicutes 門 に 大 別 さ れ る .
16SrDNA の分子系統で分類が整理された結果,最も注目
されるフローラ形成菌群は Veillonella,Acidaminococcus,
Megasphaera
など従来,グラム陰性球菌として分類さ
れていた菌群である(Table 2b)
.これらの菌種はグラ
ム陽性の Firmicutes 門の Family Acidaminococcaceae
としてまとめられている(Fig. 3 グラム陽性菌の系統
樹)
.
大 腸 フ ロ ー ラ の 優 位 菌 で あ る Eubacteria 属 菌 群 は
Actinobacteria
門の Family Coriobacteriaceae と
Firmicutes
門の Family Eubacteriaceae に分割された.
嫌 気 性 球 菌 群 で あ る Ruminococcus,Coprococcus,
および Peptostreptococcus 属の菌群は clostridia に近い配
列を持っていることから Order Clostridiales にまとめら
れ,Family Lachnospiraceae, “Peptostreptococcaceae”
,
として上位分類がまとめられている.腸内フローラとし
てはマイナーなグループであるが Sarcina 属の菌種は
Clostridium perfringens
と極めて類似した配列を保有
しており,今後も分類の再編成が必要なグループであ
る.
腸内フローラを形成する菌種は数百種類と推定されて
いるが腸内に生息する細菌種にはいまだ分離培養に成功
せず,遺伝子検査でのみ検出される菌群が多数ある.従
来の培養方法で分離培養するには膨大な労力を要してい
た(15)ので,大量の検査を行うことは困難であった.
PCR 法により,解析のスピードは従来と比較にならな
い早くなり,多くの材料を解析する環境が整ってきたた
め情報の蓄積が加速している(12, 21, 22)
.さらに,サ
ンプリングの問題から従来は口腔内と排出糞便に限られ
ていた材料が,内視鏡を使用し,小腸の十二指腸,空腸,
回腸,および大腸の盲腸,上向結腸,横行結腸などより
細かな局所から採取したデータの解析結果が報告される
ようになり,構成菌種の報告の幅も大幅に変化しつつあ
る(4, 6)
.
Firmicutes
門 に 属 す る Clostridium 属 の 菌 種 に は
Clostridium
cluster XIV と呼ばれ,しばしば優位なフロ
ーラの構成菌として遺伝子増幅では検出されるが,多数
の異なった菌群から構成されており,今後のデータの蓄
積が必要な菌群である.
PCR 法によってその存在が再認識された菌群もある.
Ruminococcus
,Coprococcus 属の菌群は嫌気性の要求
が強く,栄養要求も厳しいので,一般の微生物研究者が
培養し,分離することは困難であったのでフローラの主
要構成菌種としての認識は低かった.特異的な PCR の
primer をデザインしこれらの菌群を対象に解析すると
優位な菌群であることがわかってきた.
腸内細菌学雑誌 20 巻 3 号 2006
238
Table 1.
Taxonomic position of Bacteria and
Archaea found in human intestine
Domain
Phylum
Archaea
Euryarhaea
*
Crenarchaea
Eubacteria(or Bacteria)
Acidobacteria
Actinobacteria
Aquificae
Bacteroidetes
*
Chlamydiae
Chlorobi
Chloroflexi
Chrysiogenetes
Cyanobacteria
Deferribacters
Deinococcus-Thermus
Dictioglomi
Fibrobacteres
*
Firmicutes
*
Fusobacteria
*
Gemmatimonadetes
Nitrospira
Planctomyces
Proteobacteria
Spirochaetes
*
Thermodesulfobacteria
Thermomicrobia
Thermotogae
Verrucomicrobia
Table 2a.
Phylogenetic position of major Gram negative bacteria and Archaea found in intestine
Domain Phylum Class Order Family Genus Species Old Name Archaea Euryarchaeota Methanobacteria Methanobacteriales Methanobacteriaceae Methanobrevibacter M. smithii Archaea Euryarchaeota Methanobacteria Methanobacteriales Methanobacteriaceae Methanosphaera M. stadtmanae Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Anaerorhabdus A. furcosa 〈 -Bacteroides furcosus Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. distasonis* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. eggerthii* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. fragilis* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. ovatus* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. putredinis* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. stercoris Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. thetaiotaomicron* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. uniformis Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Bacteroides B. vulgatus* Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Porphylomonas P. gingivalis 〈 -Bacteroides gingivalis Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. denticola 〈 -Bacteroides denticola Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. disiens 〈 -Bacteroides disiens Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. oris 〈 -Bacteroides oris Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. intermedia Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. melaninogenica 〈 -Bacteroides melaninogenicus Bacteria "Bacteroidetes" Bacteroidetes Bacteroidales Bacteroidaceae Prevotella P. bivia 〈
-Bacteroides buccae, B. vivius
Bacteria "Fusobacteria" Fusobacteria Fusobacteriales Fusobacteriaceae Fusobacterium F. mortiferum* Bacteria "Fusobacteria" Fusobacteria Fusobacteriales Fusobacteriaceae Fusobacterium F. nucleatum Bacteria "Fusobacteria" Fusobacteria Fusobacteriales Fusobacteriaceae Fusobacterium F. russii Bacteria "Fusobacteria" Fusobacteria Fusobacteriales Fusobacteriaceae Fusobacterium F. varium* Bacteria "Fibrobacteres" Fibrobacteres Fibrobacteriales Fibrobacteriaceae Fibrobacter F. succinigenes 〈 -Bacteroides succinogenes Bacteria "Fibrobacteres" Fibrobacteres Fibrobacteriales Fibrobacteriaceae Fibrobacter F. intestinalis* Bacteria Spirochaetes "Spirochaetes" Spirochaetales Spirochaetaceae Treponema T. denticola Bacteria Spirochaetes "Spirochaetes" Spirochaetales Spirochaetaceae Treponema T. medium Bacteria Spirochaetes "Spirochaetes" Spirochaetales Spirochaetaceae Treponema T. multophilium Bacteria Proteobacteria Alfaproteobacteria Rhizobiales Hyphomicrobiaceae Gemmiger G. formicilis Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Citrobacter C. freundii Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Enterobacter E. cloacae Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Klebsiella K. planticola Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Klebsiella K.oxytoca Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Proteus P. vugaris Bacteria Proteobacteria Gammaproteobacteria Enterobacteriales Enterobacteriaceae Escherichia E. coli* Bacteria Proteobacteria Gammaproteobacteria Aeromonadales Succinivibrionaceae Succinovibrio S. dextionosolvens* Bacteria Proteobacteria Deltaproteobacteria Desulfovibrionales Desulfovibrionaceae Desulfovibrio D. vulgaris Bacteria Proteobacteria Epsilonproteobacteria Campylobacterales Campylobacteriaceae Campylobacter C. jejuni Bacteria Proteobacteria Epsilonproteobacteria Campylobacterales Campylobacteriaceae Helicobacter H. pylori
*species reported more than 10
腸内細菌学雑誌 20 巻 3 号 2006
240
Table 2b.
Phylogenetic position of major Gram Positive bacteria found in intestine
Domain Phylum Class Order Family Genus Species Old Name Bacteria Actinobacteria Actinobacteria Bifidobacteriales Bifidobacteriaceae Bifidobacterium B. adlescentis* Bacteria Actinobacteria Actinobacteria Bifidobacteriales Bifidobacteriaceae Bifidobacterium B. bifidum* Bacteria Actinobacteria Actinobacteria Bifidobacteriales Bifidobacteriaceae Bifidobacterium B. breve Bacteria Actinobacteria Actinobacteria Bifidobacteriales Bifidobacteriaceae Bifidobacterium B. infantis* Bacteria Actinobacteria Actinobacteria Bifidobacteriales Bifidobacteriaceae Bifidobacterium B. longum Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Atopobium A. minutum 〈 - Lactobacillus minutus Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Atopobium A. fossor 〈 - Eubacterium fossor Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Atopobium A. parvulum 〈 -Streptococcus parvulus Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Atopobium A. vaginae Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Collinsella C. aerofaciens* 〈 -Eubacterium aerofaciens Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Collinsella C. intestinalis Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Collinsella C. stercoris Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Eggerthella E. lenta* 〈 -Eubacterium lentum Bacteria Actinobacteria Actinobacteria Coriobacteriales Coriobacteriaceae Slackia S. exiguum 〈 -Eubacterium exiguum Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Anaerovibrio A. lipolytica* Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Megasphaera M. elsdenii* Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Megasphaera M. micronuciformis Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Megasphaera M. serevisiae Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Megasphaera M. micronuciformis Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Mitsuokella M. multiacida* 〈 -Bacteroides multiacidus Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Selenomonas S. ruminantium Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Sporomusa S. acidovorans Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Sporomusa S. sphaeroides Bacteria Firmicutes "Clostridia" Clostridiales Acidaminocooccaceae Veillonella V. parvula* Bacteria Firmicutes "Clostridia" Clostridiales Eubacteriaceae Eubacterium E. limosum* Bacteria Firmicutes "Clostridia" Clostridiales Eubacteriaceae Eubacterium E. biforme Bacteria Firmicutes "Clostridia" Clostridiales Eubacteriaceae Eubacterium E. cylindroides Bacteria Firmicutes "Clostridia" Clostridiales Eubacteriaceae Eubacterium E. rectale* Bacteria Firmicutes "Clostridia" Clostridiales Eubacteriaceae Eubacterium E. ventriosum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. bifermentans Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. butyricum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. clostridiiforme Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. coccoides Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. difficile Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. indolis Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. innocuum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. leptum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. limosum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. perfringens Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Clostridium C. ramosum Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Faecalibacterium F. prausnitzii* Fusobacterium prausnitzii Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Lactobacillus L. acidophilus Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Lactobacillus L. casei
Table 2b.
Continued
Domain Phylum Class Order Family Genus Species Old Name Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Lactobacillus L. mali Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Lactobacillus L. plantarum* Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Lactobacillus L. catenaformis Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Sarcina S. ventriculi Bacteria Firmicutes "Clostridia" Clostridiales Clostridiaceae Sarcina S. maxima Bacteria Firmicutes "Clostridia" Clostridiales Enterococcaceae Enterococcus E. faecalis Bacteria Firmicutes "Clostridia" Clostridiales Enterococcaceae Enterococcus E. faecium Bacteria Firmicutes "Clostridia" Clostridiales Enterococcaceae Lactococcus L. lactis Bacteria Firmicutes "Clostridia" Clostridiales Peptococcaceae Peptococcus P. niger Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Butyrivibrio B. fibrisolvens* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Coprococcus C. eutactus* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Coprococcus C. catus* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Coprococcus C. comes* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Lacnospira L. multipara Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. bromiii* 〈 -Peptostreptococcus bromii Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. albus Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. productus* 〈 - Peptostreptococcus productus Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. hydrogenotrophicus* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. lactaris* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. flavefaciens Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. hansenii* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. gnavus* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. obeum* Bacteria Firmicutes "Clostridia" Clostridiales Lachnospiraceae Ruminococcus R. torques* Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Anaerococcus A. prevotii 〈 -Peptostreptococcus prevotii Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Anaerococcus A. hydrogenalis 〈 -Peptostreptococcus hydrogenalis Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Anaerococcus A. lactolyticus 〈 -Peptostreptococcus lactolyticus Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Anaerococcus A. tetradius 〈 -Perptostreptococcus tetradius Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Finegoldia F. magnus 〈 -Peptostreptococcus magnus Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Micromonas M. micros* 〈 -Peptostreptococcus micros Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Peptinophilus P. asaccharolyticus 〈 -Peptostreptococcus asaccharolyticus Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Peptinophilus P. indolicus 〈 -Peptostreptococcus indolicus Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Peptinophilus P. harei 〈 -Peptostreptococcus harei Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Peptinophilus P. ivorii 〈 -Peptostreptococcus ivorii Bacteria Firmicutes "Clostridia" Clostridiales "Peptostreptococaceae" Peptostreptococcus P. anaerobius* Bacteria Firmicutes "Clostridia" Clostridiales Streptococaceae Streptococcus S. intermedius* Bacteria Firmicutes Mollicutes Mycoplasmatales Mycoplasmataceae Mycoplasma M. genitarium Bacteria Firmicutes Mollicutes Mycoplasmatales Mycoplasmataceae Mycoplasma M. salivarium Bacteria Firmicutes Mollicutes Mycoplasmatales Mycoplasmataceae Mycoplasma M. hominis Bacteria Firmicutes Mollicutes Mycoplasmatales Mycoplasmataceae Mycoplasma M. orale*species reported more than 10