GMV
マニュアル
おさなやすのり
∗2009 年 2 月
1
GMV
GMV はいつでも開発中なので,マニュアル中の画面イメージはそれぞれ細かいところが違って いたりしますがご容赦ください.1.1
GMV を起動するまで
Murasaki はゲノム間のアンカーを検出するためのソフトウェアで,GMV (GTK+ based Murasaki Viewer) はそれを可視化するためのツールです.
1.1.1 GMV を手に入れる
GMV は Murasaki のウェブサイト (http://murasaki.dna.bio.keio.ac.jp/gmv.html) から ダウンロードすることができます.
1.1.2 アンカーを表示する
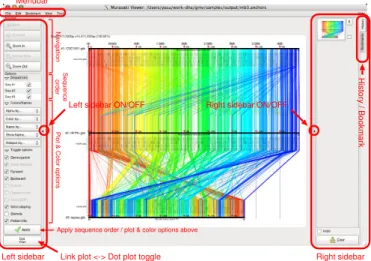
gmv を起動すると,最初に “Open Anchors File” というダイアログ (図 1) が開きますので,こ こで murasaki の出力したアンカーファイルを選択します. しばらくするとゲノム配列やアンカーが読み込まれて表示されます. 1.1.3 GMV の画面構成 図 2 が GMV のメイン画面です.いちばん大きな面積を占めているのがアンカーのプロットで, 左側にはプロットの大きさや各配列の表示 on/off など,簡単なオプションを選択するための左サ イドバーがあり,右側にはいままでの表示履歴や,あとでチェックするために bookmark (しおり) しておいた表示を一覧・呼び出しできる右サイドバーがあります.これらの機能の多くは画面上部 のメニューバーからも呼び出せます. サイドバーやメニューバーを使うと,さまざまな情報の表示・非表示を切替えることができるほ かに,プロットされている領域をマウスで操作することで,インタラクティブにさまざまな情報を 得ることができます. ∗osana@dna.bio.keio.ac.jp
図 1: .anchors ファイルの選択
Right sidebar Left sidebar
Menubar
Left sidebar ON/OFF Right sidebar ON/OFF
History / Bookmark
Navigation
Plot & Color options
Sequence
order
Apply sequence order / plot & color options above
Link plot <-> Dot plot toggle
(a) 黒い枠が出た状態でクリック
→
(b) ズーム後
図 3: 拡大表示
図 2 のプロットには CDC1551.gbk (Mycobacterium tuberclosis CDC1551 strain), leprae.gbk (Mycobacterium leprae TN strain), avium.gbk (Mycobacterium avium subsp. paratuberculosis K-10 strain) の 3 つの配列が含まれています.それぞれ,#1, #2, #3 の番号が振られており,右 側にゲノムを表す黒い太い線がその右側に引かれています.この線の左端が 1 塩基目です.この 黒い線の上下には,ORF1やリピート配列,不明な塩基など,各種のマークが付く場合があります (画面左側のオプションで指定します). ゲノムの近くにはアノテーション情報が表示されており,これには左側に小さな字でファイル名 が添えられています.ゲノム全体を表示した場合には,ひとつひとつの遺伝子は相対的にとても短 くみえるので,最初の画面ではなんとなくグレーの帯がいくつか見えている程度になります. ゲノム配列間を縦方向や斜め方向に走っている,色のついた線がアンカーです.アンカーは,一 番上の配列上での位置によって色がつけられており,左から右に向かって赤,緑,青と変化します.
1.2
アンカーやアノテーション情報のブラウズ
1.2.1 拡大表示 ゲノム全体を見ていても細かいところがわからないので,図 3 のように拡大表示をすることがで きます.プロットの上の,アノテーション等のないところ (アンカーの線が斜めになっているとこ ろ) にマウスカーソルを持っていくと,図 3 (a) のように黒い枠が表示されますので,そのままク リックするか,ドラッグしてサイズを変更してマウスのボタンを離します.すると,図 3 (b) のよ うにその部分が拡大表示されます. 拡大すると,遺伝子の向きなどがわかりやすくなったり,さらに拡大すると遺伝子の名前が表示 されたりします (図 4).遺伝子を表すグレーのブロックの,丸くなっているほうが遺伝子の終わり の部分です.正鎖にある遺伝子は右側が,逆鎖にある遺伝子は左側が,それぞれ丸くなります. 画面全体が 1 塩基幅になるまで拡大することができます.また,拡大など,表示を変更したあと で,前の表示に戻す場合には Ctrl+Z を押します. 拡大表示した状態で,表示範囲を左右に移動したり,倍率を変更したりするには,サイドバー上部にある「戻る」「進む」や「拡大」「縮小」といったボタン,あるいは Inspector (Tool → Inspector
か,Ctrl+I) を使います.
図 4: さらに拡大表示
Lock zoom ratio while varying start position
Back/Forward move stride
図 5: GMV Inspector (Zoom)
Inspector は,GMV の表示のさまざまな制御を行うダイアログで,最初に開くのは表示する範囲 を制御するための “Zoom and Navigate” タブ (図 5) です.この画面を使うと,スライダやボタン を使ったり,直接数値を入力することで移動や倍率変更などが行えます.この画面では,“Range”, “From” と “To” の 3 つのスライダを使うことができますが,“Range” の脇にある “Lock Range” をチェックしておくと,“From” や “To” を動かしたときに,表示する塩基数を維持したまま表示範 囲を変更することができます.また,下部にある “1/1”, “2/1”, . . . の選択肢を変更すると,「進む」 や「戻る」を押したときに移動する量を,現在の表示範囲に対する割合で選択することができます. 1.2.2 アノテーション情報を参照 GMV では,GenBank ファイルや,その他のファイルからアノテーション情報を読み込んで表 示することができます.今回の実習で使っている配列は GenBank ファイルなので,アンカーを読 み込んで表示したときに,同時にアノテーション情報も表示されています. 基本的な情報を参照 ゲノム全体を表示しているときには遺伝子は短くてほとんど見えませんが, 拡大するとひとつひとつの遺伝子を表すマーカーをはっきり見ることができます.遺伝子の名前の 表示・非表示はある程度自動的に切り替わりますが,サイドバーの “Show Name...” のメニューを 使うと,常に表示したり,逆に常に表示しなかったりといったオプションを選択することができま す.選んだだけでは反映されないので,“Apply” を押してください. マウスを遺伝子マークの上に持って行くと名前が表示されますが,クリックするとより詳しい情 報を表示するウィンドウ (Gene Information Dialog) が開きます.ここでは,クリックした遺伝子
図 6: Gene Information Dialog (Browse)
のほかに,その遺伝子にアンカーでつながっている遺伝子の情報もみることができます.この際, 表示は図 6 のようになり,メインのウィンドウでは注目している遺伝子およびそれとつながってい るものに赤いマークがつき,Gene information dialog ではそれらの遺伝子の一覧が表示されます.
Gene information の下部にはいくつかのオプションがあり,マウスをクリックしなくても常に マウスポインタの位置にある遺伝子を表示する (Follow to Mouse),アンカーでつながっている遺 伝子を表示する・しない (Show connected genes) などを選ぶことができます.
また,アノテーション情報に遺伝子の機能に関する長めの注釈 (Note) や転写後のアミノ酸配列 (Translation) などが含まれている場合,それも表示されますが,長い場合には “MIGNGGAGGS-GAPG. . . ” のように,省略して表示されます.このとき,“Note” や “Translation” をクリックす ると全体を表示することができます (図 7).また,アミノ酸配列などの情報はマウスで選択して右 クリックすると,コピーしてほかのソフトウェアに貼り付けることができます.
データベースを参照 遺伝子マークを右クリックしたり,Gene information dialog に表示されて
いる遺伝子情報の “DB Links” のプルダウンメニューを使うと,NCBI などのデータベースを参照 することができます.ただし,“Open All” などを使うと,一気にたくさんブラウザのウィンドウ が開くことになりますので,注意しましょう.
遺伝子を検索 Gene information dialog の上の方にあるタブを “Browse” から “Search” に切り替
えると,アノテーション情報を文字列検索することができます. 図 8 (a) が検索画面です.上の方に検索文字列を入れるテキストボックスがあり,その下で検索 対象の配列を選びます.また,大文字小文字の区別をなくすには,“case insensitive” をチェックし てください.“Query” をクリックする (または Alt+q) と,検索が実行されます. 検索結果はこのウィンドウの下半分に表示されますが,一度に表示されるのは 50 件までですの で,それを超える場合には単にスクロールするだけでなく,「戻る」や「進む」を使用してページを 切り替えてください.また,検索された遺伝子にはプロット中で赤いマークが付きます (図 8 (b)). 検索結果から,その遺伝子周辺のプロットに切り替えるには,検索結果にある “Go” ボタンを使 用すると便利です.
(a) 配列の一部を表示 →
(b) 配列全体を表示
図 7: アミノ酸配列の表示
(a) アンカーや遺伝子をクリック
→
(b) 位置調整後
図 9: 拡大表示
(a) All anchors
→ (b) Turn #1 → (b) Turn #2 図 10: 画面に入りきらない場合 1.2.3 遺伝子やアンカーを基準にして配列をずらす 拡大表示にすると,見たいアンカーの先が画面の外に行ってしまう場合があります.このような 場合には,配列を表す黒い線の近くで (拡大表示するための黒い枠が表示されていないときに),遺 伝子やアンカーをダブルクリックすると,そのアンカーを基準にして配列をずらして表示すること ができます (図 9).ただし,ある配列では同じ位置に複数のアンカーがあって,それらが別の配列 では遠く離れた位置にある,というような場合にはうまく画面に収まらない場合があります. このような場合は,マウスをその位置のまま移動せずに続けてダブルクリックすると,1 画面に 入る範囲で順番に,その遺伝子を通るアンカーを表示することができます (図 10).ダブルクリッ クを続けると,「すべてのアンカーで位置あわせ」→「いくつかのアンカーで画面に収まる範囲で 位置あわせ (1 回目)」→「残りのアンカーのいくつかで画面に収まる範囲で位置あわせ (2 回目)」 のように,該当する遺伝子を通るアンカーをすべて表示し終わるまで順次アンカーを表示していき ます.この場合,画面下部に,“Justification mode:” と表示され,現在の表示が何ステップ目なの かを知ることができます. これにより,どういった遺伝子をアンカーがつないでいるかを把握することができます. また,画面の右端のほうにある遺伝子やアンカーを画面中央に持って行きたい場合は,右クリッ クして “Align & center this gene” や “Align & center this anchor” を選択します (図 11).
ずらして表示した配列を元に戻すには,“View” → “Justify Left” を選択するか,Ctrl + < を 押します.gmv のデフォルトは左揃えですが,Justify Right や Justify Center を選べば右揃えや 中央揃えにすることもできます.
図 11: 遺伝子を右クリックしてメニューを表示
1. Reorder the sequences by drag & drop
2. Click "Apply" to redraw
図 12: ゲノム配列を並べ替え
1.2.4 ゲノム配列の順序を変更する
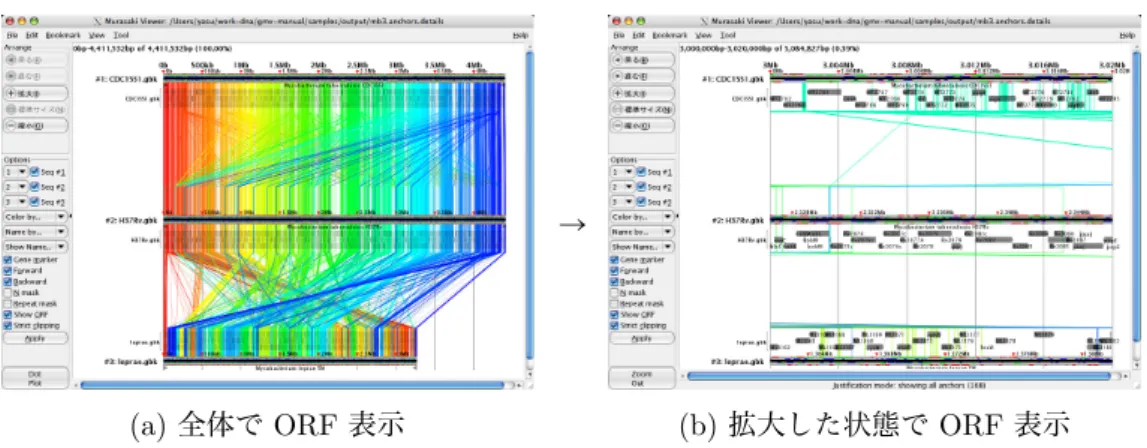
GMV では,Murasaki を実行したときに与えられた順番で配列を表示しますが,そうでない順番 に並べ変えて表示したい場合もあります.このような場合には図 12 のように,左サイドバーの中程 にある “Sequences” の欄をドラッグ& ドロップで並べ替え,左サイドバーの下の方にある “Apply” ボタンを押すことで,希望する順番に並べ替えることができます. また,“Sequences” の欄にあるチェックボックスを外すと,そのゲノム配列を非表示にすること もできます. 1.2.5 ORF を表示する 最初の状態では配列データが読み込まれていないために,サイドバーの “Show ORF” などを使っ て ORF を表示することができません.配列データを読み込むためには,サイドバー上部の “Load Sequences to show ORFs” ボタンを押します (このボタンが表示されていない場合には配列データ
(a) 全体で ORF 表示
→
(b) 拡大した状態で ORF 表示
図 13: ORF 表示
が読み込まれており,ORF などを表示することができます).一旦配列が読み込まれれば,サイド バーの「Show ORF」を on/off して「Apply」を押すことで,ORF の表示・非表示を切替えるこ ともできます.
ORF は,ゲノムを表す横棒の上下にそれぞれ 3 フレームずつ,赤・緑・青で表示されます (図 13).上側の 3 本が正鎖上の ORF,下側の 3 本が逆鎖上の ORF です.ゲノム全体を表示している と,ORF はとても短いので見づらくなりますが,拡大表示にすると見やすくなります.
ORF を表示せず,配列を読み込むだけにしておきたい場合は,“File” → “Load Sequences” を 選択します.既に配列が読み込まれている場合,このメニューは選択できません. 1.2.6 過去の表示履歴を参照する 右サイドバーには,ファイルを開いてからいままでの表示履歴が縮小表示されます.これらの 表示履歴はボタンになっており,ボタンを押すことでその画面を呼び出せます.履歴表示できる数 には限りがあるので,重要なところはボタン右のチェックボックスをチェックしておくと,古い表 示を破棄する際に残しておくことができます.また,チェックが入っていないボタンは,右サイド バー下の Clear ボタンを押せば,削除することができます. 1.2.7 現在の表示状態を記憶する メニューバーの “Bookmark” を使うと,現在の表示状態を記憶しておくことができます.いく つかの遺伝子を探すような場合に,場所をメモリしておくのに便利です.“Bookmark” → “Add Bookmark” を選択するか,あるいは Ctrl+D を押すと,図 14 (a) のようなダイアログが出ます. ここでいま表示している画面の場所に名前をつけておくと,あとで “Bookmark” メニューや右サイ ドバー (図 14 (b)) から探すことができます.右サイドバーの縮小表示されたイメージは表示履歴と 同様ボタンになっており,これを押すことでその画面を呼び出すことができます.また,Bookmark につけた名前は,ボタンの上でしばらくマウスを静止させると図 14 (b) のように表示されます.
Bookmark は GMV を終了するときや,“Bookmark” → “Save Bookmarks Now” を選んだとき に保存されます (後者の操作で手動で保存していれば,GMV がクラッシュした場合もたいてい大 丈夫です) ので,レポートの作成などに活用してください.
(a) Ctrl+D でブックマーク追加 (b) 右サイドバーの表示 図 14: Bookmark の使い方例 図 15: GMV によるドットプロット 1.2.8 プロットを保存する 画面に表示されているプロットを画像ファイルとして保存するには,“File” から “Save PNG” を 選択します.“Save PDF” もありますが,1GB 以上の巨大な PDF ファイルができる場合がありま すので,なるべく使わないようにしましょう. 1.2.9 ドットプロットへの切り替え GMV のデフォルトの表示では,すべての配列を横向きに並べて,その間をアンカーで接続する プロット (リンク図,と呼んでいます) になりますが,Ctrl+T (もしくは,画面右下の “Dot Plot” ボタン) で 2 種のゲノム間の関係を表す古典的な方式であるドットプロット (図 15) に切り替える こともできます.この表示では,さきほどの画面の一番上の配列を横に,残りの配列を縦にとって, アンカーのあるところを表示します.まったく同じ配列ならば,左下から右上に向かってまっすぐ な線が一本引かれるわけですが,実際にはそうはなりません.配列のどの部分がゲノム間で同じ向 き (左下から右上方向) だったり,逆向き (左上から右下方向) だったり,あるいは同じ配列が繰り 返し出現している様子 (縦や横の同じ位置にマークが並ぶ) などといったことが読み取れます.
(a) すべてのアンカーを表示 → (b) フィルタを使用 図 16: tf-idf フィルタの適用 この画面では,以降で説明するようなインタラクティブな機能の多くは利用できません.もとの 表示に戻すにはもう一度 Ctrl+T か,サイドバー下部の “Chrom Links” ボタンを押します.
1.3
フィルタ・レポートの利用
1.3.1 tf-idf スコアでフィルタを行うInspector の,“tf-idf Anchor Filter” タブ (図 16) を使うと,tf-idf スコアを用いてアンカーを フィルタすることができます.このタブにはふたつのスライダがあり,それぞれ残すアンカーのス コアの上限・加減を設定することができます.非表示になるスコアは,ヒストグラム上でグレーに 表示され,残るアンカーの数の概算値がスライダーの下に表示されます.スコアの高いアンカーだ けを残してやると,よく保存されている領域が残ります.
1.3.2 アンカーの有無で遺伝子をフィルタ
アンカーの有無で遺伝子をフィルタするには,Inspector の “Annotation Tracks and Filters” タ ブ (図 17) を選択します.
このタブは,基本的には表示するアノテーションのファイルを追加・削除するためのものですが, 一番下の “Hide unanchored genes” をチェックして「適用」すると,アンカーが通っていない遺伝 子を消したり,その逆の “Hide anchored genes” によってアンカーのない遺伝子だけを表示したり することができます.tf-idf フィルタと組み合わせて使用すると,種間での遺伝子ごとの配列保存 度を知るのに役立ちます.
また,スコアのついたアノテーション情報 (発現情報など) が読み込まれている場合,このタブ を用いて,アノテーションのスコアによる遺伝子やアンカーのフィルタを設定することも可能です.
図 17: GMV Inspector (Annotation Tracks and Filters)
図 18: レポート画面
1.3.3 統計情報など
アンカー・塩基数統計 アンカーに関する統計は “Tool → Sequence statistics (Ctrl+R)” で見られ
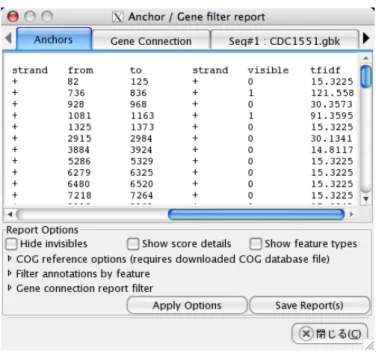
ますが,最初はアンカーの数しか表示しないので,細かな情報を見るにはそこで “Generate Details” というボタンを押してやる必要があります. 配列が読み込まれて,ORF などを表示できる状態であれば,もうすこし詳しい情報が表示され ますが,“Generate Details” ボタンが表示されている場合にはまだ追加の表示項目が計算されてい ないということですので,押してみてください. 統計情報は HTML (Internet Explorer などで表示するもの) またはプレインテキスト (ふつうの テキストファイル) の形で保存することができます. 遺伝子の接続性・フィルタレポート “Tool → Reports” では,どの遺伝子とどの遺伝子がアンカー
でつながっているかの一覧 (gene connection report) と,フィルタで残った遺伝子の一覧などのレ ポートを見ることができます.これも,スコアの計算をいろいろやる必要があるので,表示までは しばらく時間がかかる場合があります (図 18).
Anchors それぞれのアンカーの開始点 (from),終了点 (to),方向 (strand) とフィルタの結果 (visible: 1 なら表示,0 なら非表示) を表示.tf-idf スコアが読み込まれている場合にはそれ (tfidf) を表示.スコアのついたアノテーション情報が読み込まれている場合には,アノテー ションのスコアから算出されるアンカーのスコアも表示される. Gene connection それぞれの配列のアノテーション間で,アンカーで接続されているものの一 覧.N/A になっているものは,他の配列についているアノテーションに対応する場所にアノ テーションがないことを示す.複数のアノテーションファイルが読み込まれている配列では 最初のファイルのみを参照する.
Seq#x: (file) 各アノテーションファイルに含まれるアノテーション情報の開始点 (from), 終了点
(to), 方向 (strand) とフィルタの結果 (visible) と,含まれるアンカーの数 (#ancs) を表示. tf-idf スコアが読み込まれている場合には,tf-idf スコアから算出される各アノテーションの スコアを,スコアのあるアノテーション情報が読み込まれている場合にはそのスコア (およ びそれから算出される,同じ配列に割り当てられている別のアノテーション情報についての スコア) を表示.
このレポートダイアログにはいくつかのオプションがありますが,“Show Score details” は,た とえば遺伝子に含まれるアンカーすべての tf-idf スコアを列挙するようなことをするため,場合に よってはレポートのサイズが巨大になりますので注意してください. このレポートは,CSV 形式 (タブ区切りファイル) で保存することができます.