バイオインフォマティクス
実習テキスト
リンク集
http://www.cbrc.jp/~hirokawa/toyama.html
産総研生命情報工学研究センター
広川貴次
ゲノム情報処理工学・実習テキスト
目次
1.遺伝子配列・アミノ酸配列を取得する ... 3 2.ホモロジー検索 ... 8 3.PCR のプライマーを設計する ... 12 4.インターネットを利用したタンパク質立体構造予測 ... 15 5.複数配列のアラインメント ... 20 6.配列解析による標的タンパク質同定 ... 22 7.統合データベースを用いた標的タンパク質同定 ... 28 8.タンパク質立体構造に基づく機能推定 ... 34 9.タンパク質立体構造に基づく酵素活性部位予測 ... 37ゲノム情報処理工学・実習テキスト
1.遺伝子配列・アミノ酸配列を取得する
【概 略】 代表的な分子生物学データベースの多くは、セントラルドグマで記述されるような階層 に沿って分類することができます。分類は大きく DNA、RNA 配列などの塩基配列データ ベース、アミノ酸配列データベース、タンパク質立体構造データベースに分けらますが、 データベース開発の歴史的背景や管理運営の拠点、フォーマットの独自性から、塩基配列 データベースを見ても3つのデータベース(DDBJ、GenBank、EMBL)が存在します。 最近では、登録されている遺伝子やタンパク質は、同じ階層分類にあるデータベース間で はほとんど重複していますが、階層間の登録数には大きな違いがあります。特にタンパク 質立体構造データベースは、原子座標決定にX 線や NMR などの実験技術を用いているた め、塩基配列やアミノ酸配列に比べると登録数が少ないのが現状です。 【操作1】GenomeNet を使った配列取得 GenomeNet システムを使って配列情報を取得します。例題としてヒト上皮成長因子受 容体遺伝子を用います。最初にタンパク質アミノ酸配列データベース UniProt で検索 し、その後、DR 項目を使って GenBank や PDB のエントリーを調べます。 GenomeNet にアクセスします。http://www.genome.ad.jp/
GenomeNet のホームページ上部にある”Search”リストからタンパク質アミノ酸配列 データベース”UniProt”をクリックしますゲノム情報処理工学・実習テキスト 図 UniProt の選択 上皮成長因子受容体遺伝子のキーワードである”EGFR”と生物種を特定するための” human”を検索フォームに入力します。通常、限定したキーワードを用いない限り、 検索結果は、いくつかの候補を出力します。例えば、今回の場合は、VEGFR(血管内 皮増殖因子受容体)が含まれていますので、タイトルを確認して目的の遺伝子を示す エントリーを選択することが重要です。また UniProt 検索結果は、塩基配列データベ ースEMBL からタンパク質コード領域をアミノ酸配列に変換した TrEMBL データベ ースにも検索を実行しています。tr:で始まるエントリーは、TrEMBL に対する検索結 果となります。sp:で始まるエントリーは、SwissProt に対する検索結果となります。 EGFR 検索結果
ゲノム情報処理工学・実習テキスト データベース内容を確認します。ここでは、検索結果より”sp:EGFR_HUMAN”を選 択します。データベースには、登録日や配列、アノテーション更新日、酵素番号や関 連論文など記載されています。アミノ酸配列情報は、ページの最後にあります。先頭 にある”SQ”をクリックすると一行目に”>”で始まるタイトル行と、二行目以降に アミノ酸配列の一文字表記が連なったテキストが表示されます。この書式をFASTA 形 式と言います。いろいろな解析の基本となる重要な入力形式です 図 FASTA 形式 データベースの DR 行目を利用して、他のデータベース上の関連エントリーを調べる ことができます。塩基配列情報は、GenBank や EMBL、立体構造情報は PDB をクリ ックします。関連エントリーは、単一でない場合があります。塩基配列の場合、mRNA としてのエントリーもあれば、任意のエクソン領域のエントリーもあります。立体構 造情報でも、実験手法の違いやリガンド結合状態、変異体などによって同一遺伝子で も複数のエントリーが存在します。各エントリーの記述を確認して利用目的に合った エントリーを選択することが重要です。 図 他のデータベースへのリンク情報を示すDR 項目
ゲノム情報処理工学・実習テキスト 【操作2】ゲノム種別配列の取得 NCBI の FTP サイトより例題として枯草菌ゲノムの配列情報を取得します。ユーザー インターフェースを使って個々の遺伝子を検索したときと違い、ここでは枯草菌ゲノ ムの全配列データの取得を行います。 Web 経由で NCBI の FTP サイトにアクセスする
http://www.ncbi.nlm.nih.gov/Ftp
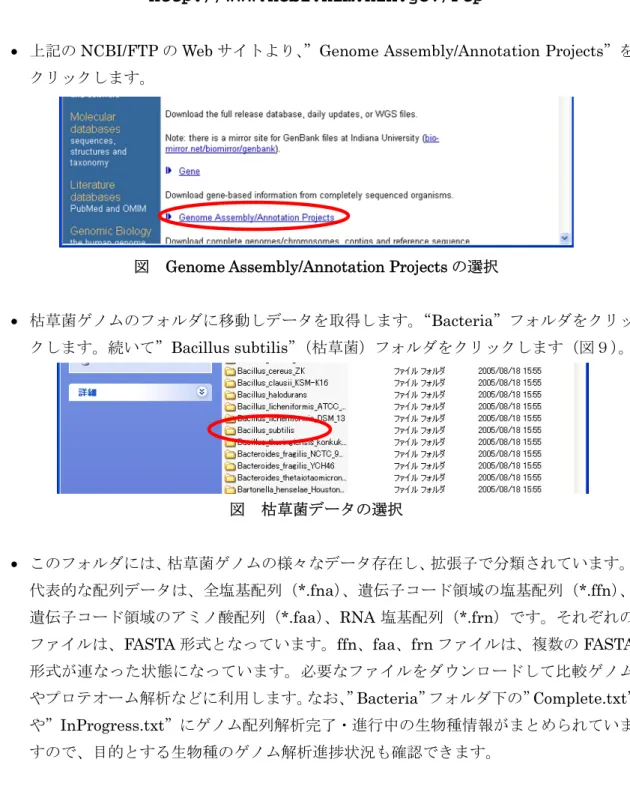
上記の NCBI/FTP の Web サイトより、”Genome Assembly/Annotation Projects”を クリックします。
図 Genome Assembly/Annotation Projects の選択
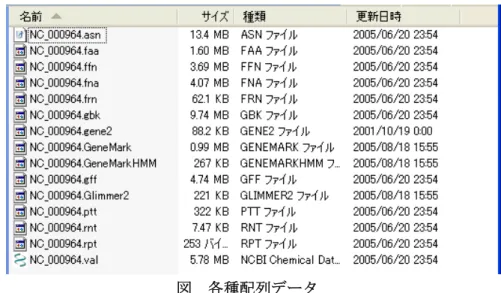
枯草菌ゲノムのフォルダに移動しデータを取得します。“Bacteria”フォルダをクリッ クします。続いて”Bacillus subtilis”(枯草菌)フォルダをクリックします(図9)。 図 枯草菌データの選択 このフォルダには、枯草菌ゲノムの様々なデータ存在し、拡張子で分類されています。 代表的な配列データは、全塩基配列(*.fna)、遺伝子コード領域の塩基配列(*.ffn)、 遺伝子コード領域のアミノ酸配列(*.faa)、RNA 塩基配列(*.frn)です。それぞれの ファイルは、FASTA 形式となっています。ffn、faa、frn ファイルは、複数の FASTA 形式が連なった状態になっています。必要なファイルをダウンロードして比較ゲノム やプロテオーム解析などに利用します。なお、”Bacteria”フォルダ下の”Complete.txt” や”InProgress.txt”にゲノム配列解析完了・進行中の生物種情報がまとめられていま すので、目的とする生物種のゲノム解析進捗状況も確認できます。
ゲノム情報処理工学・実習テキスト
ゲノム情報処理工学・実習テキスト
2.ホモロジー検索
【概 略】 進化的に共通な祖先を持つ遺伝子・タンパク質間の関係をホモログといいます。ホモロ ジー検索とは、ある任意の配列に対するホモログ配列を塩基配列もしくはアミノ酸配列デ ータベースから配列類似性の尺度に基づいて検索する技術です。ホモロジー検索のような 配列比較は、配列-構造-機能の関係を調べる基本的な解析の一つで、特に構造や機能が 未知の遺伝子・タンパク質は、構造・機能既知の配列データベースに対してホモロジー検 索を行うことで、機能や構造を推定することができます。 通常、問い合わせ配列は、塩基配列かアミノ酸配列で表現され、問い合わせ先となるデ ータベースは、チュートリアル1で紹介した、DNA、RNA 塩基配列データベース(DDBJ、 GenBank、EMBL)やタンパク質アミノ酸配列(UniProt、PIR、PRF)・立体構造データ ベース(PDB)になります。配列類似性の比較は、塩基配列でもアミノ酸配列でも可能で すが、アミノ酸配列レベルでの比較が有効とされています。DNA 配列に起こりうる変異に はアミノ酸配列に変化を与えないもの(Synonymous)があることや、4つの塩基で表現さ れる塩基配列より20種のアミノ酸残基によるアミノ酸配列を用いた配列比較の方がより 明確に進化的関係を表すことができるからです。また、6フレーム変換を用いることでDNA 配列が問い合わせ配列の場合でも、タンパク質アミノ酸配列・立体構造データベースに対 してホモロジー検索できます。ただし、DNA 配列に非コード領域が含まれていないかなど 予め確認が必要です。 【操作1】BLAST(Web サーバー)を使った配列検索 NCBI サイトの BLAST を使って、ヒト上皮成長因子受容体遺伝子 EGFR のアミノ酸 配列を問い合わせとして検索を行います。EGFR HUMAN の配列取得は、チュートリ アル1を参考にするか、下記のサイトを参照ください http://www.genome.ad.jp/dbget-bin/www_bget?sp:EGFR_HUMAN NCBI の BLAST にアクセスします http://www.ncbi.nlm.nih.gov/BLAST/ この実習では、アミノ酸配列を問い合わせ配列として、タンパク質アミノ酸配列・立 体 構 造 デ ー タ ベ ー ス に ホ モ ロ ジ ー 検 索 を し ま す の で 、”Protein-protein BLAST (blastp)”をクリックします。
ゲノム情報処理工学・実習テキスト 図 blastp の選択 ブラウザの編集機能を使って EGFR_HUMAN のアミノ酸配列をコピーし、BLAST ペ ージの”Search”カラムにペーストします。”Choose database”は、”nr”の設定にしてお きます。nr とは、非冗長のアミノ酸配列データベースセットで、GenBank CDS translations、RefSeq Proteins、PDB、SwissProt、PIR、PRF から集められていま す。”BLAST!”ボタンをクリックし、検索を実行します。 図 BLAST の実行 検索が実行されると、最初にタンパク質ドメインの検索結果が出力されます(これは NCBI サイトの BLAST 独自のオプションです。ドメイン情報は構造や機能を推定する ことに活用できます。)。BLAST の結果は、”Format!”をクリックすると別ウィンドウ
ゲノム情報処理工学・実習テキスト が起動し結果が出力されます。 図 ドメイン検索結果 出力の最初では、検索でヒットした配列と問い合わせ配列間のアラインメントスコア と領域が視覚的に表現されています(図4)。これはクリッカプルマップになっていま すので、興味のあるヒットを選択するとアラインメント情報へジャンプします。 図 ヒット結果の表示 続いてヒットした配列のリストが出力されています。Socre や E-value のほか、”G”や”S” といったアイコンがリストの右側に付加されています。これらは、Entrez(第2部- 1の概説参照)のGene データベースと Structure データベースへのリンクを意味して います。リストの先頭箇所をクリックするとヒットした配列の詳細情報を調べること ができます。また Score 箇所をクリックすると問い合わせ配列とヒットした配列のア ラインメント情報へジャンプします。
ゲノム情報処理工学・実習テキスト 図 結果の詳細 アラインメント結果は、上段に問い合わせ配列、下段にヒット配列、中段に一致度に 関する情報で表現されています。中段では、上段と下段で文字が一致している場合は、 その文字が記され、一致していない場合は空白となっています。ただし、文字の不一 致でもアミノ酸残基の性質が似ているもの同士であれば、+が記されています。 図 アラインメントの詳細 問い合わせ配列には、X でマスクされる領域が存在することがあります。これは BLAST のフィルタリングが働いていることを意味しています。このフィルタリングは、 Proline-rich 領域などの繰り返し配列を検索前にマスクする機能をもっています。これ により有意でない一致がスコアに影響を与えないようになっています。
ゲノム情報処理工学・実習テキスト
3.PCR のプライマーを設計する
【概 略】 遺伝子解析の基盤技術である PCR(ポリメラーゼ連鎖反応)において、プライマーの設 計は、伸長効率が良く、特異性が高いPCR を実現するための重要な課題の一つです。コン ピュータによるプライマー設計ツールとは、入力配列(DNA 配列)に対して、プライマー 長、伸長サイズ、GC 含量、オリゴヌクレオチド融解温度(以下、Tm 値)などのパラメー タを調整しながら候補となる最適なプライマーを出力するものです。プライマー設計ツー ルによっては、複数配列を入力してマルチプルアラインメント(第2部-5)の結果から 保存度の高い領域を同定し、プライマー設計に利用するものもあります。プライマー設計 ツールは、PCR 以外にも DNA シークエンシングやクローニングにも利用されています。 設計されたプライマーは、BLAST などのホモロジー検索ツールも併用して特異性を調べる のも良いでしょう(問い合わせ配列長が短いので検索結果の判断には注意が必要です)。 【操作1】p53 配列の取得 チュートリアル 1 を参考に GenomeNet の Search カテゴリから GenBank を選択しま す。検索フォームに、癌抑制遺伝子p53 のデータベース ID である”BC003596”を入力 し検索します。データベース中に”ORIGIN”という配列情報の項目があります。これを クリックすると塩基配列情報のみ表示されますので、これをコピーしておきます。
【操作2】Primer3 による解析
Primer3 の Web サイトにアクセスします。操作 1 で取得した入力配列は、ヒト由来で あるため、”Misprinting Library (repeat library)から”HUMAN”に設定します。つづい て、操作1 でコピーした p53 の配列を入力フォームにペーストします。
図 配列の入力
Primer3 のパラメータは、適宜、デフォルト設定値を参考にして調整します。ここで はデフォルト値のまま実行します。もし結果を見て、より多くの候補プライマーをリ ストアップしたい場合には、各パラメータの上限、下限値の調整の他、”Product Size
ゲノム情報処理工学・実習テキスト
Range”に新たなサイズ範囲の追加や、”Number of Return”の値を高く設定して再実行 します。
図 パラメータ設定領域
検索結果として Primer3 の出力のヘッダには、タイトルが表示されます(タイトル名 は、FASTA 形式の”>”行の内容や”パラメーラ項目の”Sequence ID”に入力した情報が 反映されます)。続いて、検索結果として、候補プライマーから選ばれた最適なプライ マーの結果がTm 値などの情報とともに表示されます。
図 検索結果
これは、パラメータ設定時に指定した”Opt”値に近いものや”Product Size Range”の優 先 順位 が反映 され ていま す。 さらに プラ イマー 箇所 は、入 力配 列上に”>>>>>>”
ゲノム情報処理工学・実習テキスト や”<<<<<<”で示されています。その他の候補プライマーは、”ADDITIONAL OLIGOS” 項目に列挙されています。検索結果の末尾には、”Statistics”項目があり、どのパラメ ータ項目でプライマーが除外されたかが確認できますので、この情報を参考しパラメ ータ調整を行ってもよいでしょう。 図 検索結果詳細
ゲノム情報処理工学・実習テキスト
4.インターネットを利用したタンパク質立体構造予測
【概 略】 タンパク質立体構造予測とは、立体構造未知のタンパク質に対して配列情報から計算機 的手法により立体構造を予測するもので、最近では、予測精度の向上や、便利なウェブツ ールが公開されていることから、タンパク質立体構造予測を利用するバイオ実験研究者も 増えてきています。代表的なタンパク質立体構造予測法として、構造既知のタンパク質(鋳 型タンパク質)との配列相同性(30%以上)に基づいて予測するホモロジー(比較)モデ リング法が挙げられます。一般に配列相同性が高ければ、予測される構造の信頼度も高く なります。ここでは、インターネットを利用して比較的簡単に利用できるSWISS-MODEL を紹介します。構造構築後の極小化計算などが簡易に行われているため、例えば、折りた たみ構造の確認や部位特異的変異箇所の解析など、厳密なモデルを必要としない解析に向 いています(First Approach Mode の場合)。【操作1】SWISS-MODEL へのアクセス
SWISS-MODEL の Web サイトにアクセスします。
http://swissmodel.expasy.org/SWISS-MODEL.html
画面左側にある Menu の”First Approach Mode”をクリックします。
図 SWISS-MODEL のトップページとメニューの選択
【操作2】配列の入力と実行
ゲノム情報処理工学・実習テキスト
もの)を入力します。そして対象タンパク質のアミノ酸配列(一文字表記)を入力(ペ ースト)します。入力が完了したら”Submit Modelling Request”をクリックします。
図 入力項目 しばらくすると予測結果が表示されます。問い合わせ配列のどの部分が構造予測できた を示す図に続いて、鋳型となったタンパク質の情報が表示されます。図の場合は、 PDB-ID で 1qtq というエントリーの A 鎖を鋳型としたことになります。構造予測が終 了した知らせやアクセスのためのパスワードなどは、入力項目で入力した電子メールア ドレス宛にメールが届きます。 図 1 予測結果画面 【操作3】予測構造のダウンロード
ゲノム情報処理工学・実習テキスト
PDB 形式でデータをダウンロードする場合には、download model 行にある”as pdb”を クリックし、データを保存します。 図 予測構造のダウンロード 予測結果のページには、構造予測に用いた入力配列と鋳型タンパク質間のアラインメ ントも表示されています。この情報は、鋳型タンパク質で機能部位が明らかな場合、 予測したタンパク質のどの部位に対応するのか、またアラインメント情報にさかのぼ って予測構造を修正したい場合などに必要となります。 ホモロジーモデリングで予測した構造の精度は、鋳型タンパク質との配列相同性に依存 してきます。SwissModel_TraceLog ファイルを確認して、予測した構造が鋳型タンパ ク質とどのくらいの配列相同性に基づいて構築されたものかをきちんと把握しましょ う。例えば、予測したタンパク質構造と低分子の相互作用解析などを目的とする場合は、 約 90%以上の配列相同性に基づいたホモロジーモデリングにより得られた構造が必要 だといわれています。 【操作4】予測構造の評価 SWISS-MODEL サーバーでは、予測した構造に対する評価を行うことができます。 Anolea/Gromos/Verify3D の 3 種類の評価法を選択することができます。以下、Verify3D を用いた予測構造の評価を行います。Verify3D では、解像度の高い代表的な結晶構造 データセットに基づく統計的な解析から、天然構造に見られる残基毎のタンパク質内 部への埋没度合いと周りをとりまく極性環境、非共有結合のパターン等を指標化し、 実際の予測構造がその指標にどれだけ則しているかによって予測構造の質を評価する ことができます。デフォルトで on になっている anolea を off に変更後、verify3d を on にして”show”ボタンをクリックします。
図 Verify3D の選択
ゲノム情報処理工学・実習テキスト 示されます。3D-1D プロファイルは、値が高い領域ほど、統計的な天然構造の折りた たみ環境(アミノ酸残基の内部構造への埋没度合いと周りの極性環境)に近いことを 示しています。プロファイル値が 0 以下については、折りたたみ状態が適切ではない ことを示唆しています。 図 Verify3D の解析プロット 縦軸のプロファイル値が 0 を下回っている場合は、対応する位置の SWISS-MODEL の出力ファイルの中の鋳型タンパク質とのアラインメントを修正し再度モデル構築を 行うことで改善されることがあります。他の改善策として、分子力学・分子動力学計 算を用いて最適化構造を構築する方法もありますが、計算機資源が必要なことや専門 的な知識も必要とするため、中・上級者向けとなります。 【操作5】Workspace の活用 SWISS-MODEL では、一度解析を行った際に、電子メールアドレスをユーザー名とし たWorkspace と呼ばれるアカウントのようなものが作成されます。過去のジョブの表 示や次回の構造予測などは、Workspace へログインして行うことができます。ログイ ンのためのパスワードは、最初に解析した際に、メールで連絡がありますので、大切 に保存してください。ログインは、モデリングページのヘッダ部分から行えます。
ゲノム情報処理工学・実習テキスト
ゲノム情報処理工学・実習テキスト
5.複数配列のアラインメント
【概 略】 配列アラインメントは、ホモロジー検索のように2つの DNA・RNA もしくはタンパク 質間で配列類似性(ペアワイズアラインメント)を調べる目的だけではなく、例えば、特 定の遺伝子ファミリーについて複数配列間のアラインメントを行うこと(マルチプルアラ インメント)で共通に保存されている配列領域を発見することもできます。このような配 列領域は「配列モチーフ」と呼ばれ、機能や構造について重要な知見を与えます。また、 遺伝子の系統樹を作成する時にもマルチプルアラインメント結果が利用されています。複 数配列をアラインメントする場合にも動的計画法が基本となりますが、多次元の動的計画 法が計算量の問題から現実的に困難であるため、ペアワイズアラインメントを積み上げ式 に実行するようなツリーベース法や反復改善法などの手法が用いられています。 【操作1】Clustal X によるマルチプルアラインメントの実行 ローカルマシンで実行できる Clustal X を用いたマルチプルアラインメントの実行方 法を紹介します。ここでは、Windows 版を例に紹介しますが、Macintosh 版でも同様 の手順となります。以下のサイトからClustal X を取得します。http://bips.u-strasbg.fr/fr/Documentation/ClustalX
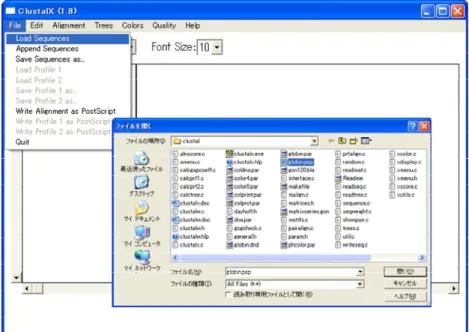
/ バージョンやプラットフォームを確認後、ファイル(zip ファイル)をダウンロードし ます。ここでは、Windows 環境で稼動する clustalx1.8.msw.zip をダウンロードし、 WinZip など(ファイル圧縮・解凍ソフトウェア)を利用して解凍します。ファイルの 解凍先のフォルダ名には、日本語が含まれないように注意してください。解凍後は複 数のファイルが展開されますが、その中にclustalx.exe という実行形式のファイルがあ ります。このファイルをクリック(もしくは、コマンドプロンプトからの実行)する とClustal X が起動します。 File メニューから”Load Sequences”を選択します。ここではチュートリアル用に ClustalX パッケージに用意されている globin.pep を入力ファイルに指定します。 Clustal X では多くの入力形式に対応していますが、各自で入力ファイルを用意する場 合には、FASTA 形式が最もよく利用されています(globin.pep は、NBRF/PIR 形式と 呼ばれるFASTA 形式に似たファイル形式で記述されています)。
ゲノム情報処理工学・実習テキスト
図 Clustal X の起動とファイルの読み込み
globin.pep を読み込むとアラインメントされていない7つのタンパク質配列が含まれ ていることがわかります。マルチプルアラインメントの実行は、Alignment メニュー から”Do Complete Alignment”を選択します。出力ファイル名を確認後、”ALIGN”ボ タンをクリックすると計算が実行されます。Clustal X では、アラインメント結果の他 に、各アラインメント位置における保存性を反映させたスコアがウィンドウの下部に グラフ表示されます。
ゲノム情報処理工学・実習テキスト
6.配列解析による標的タンパク質同定
【概 略】 タンパク質配列の機能を推定する場合、BLAST のような配列相同性に基づく方法の他に モチーフ検索や膜タンパク質予測、局在予測などがあります。これらは、配列相同性検索 などでは判断することが困難な場合に非常に役に立ちます。例えば、モチーフ検索結果で キナーゼによく存在する機能モチーフが見つかれば、対象とする配列がキナーゼドメイン である可能性が示唆されますし、結果として抗がん剤標的タンパク質と考えることができ るかもしれません。またシグナルペプチド予測の結果、シグナルペプチドが推定された場 合、分泌タンパク質だと考えることができます。膜タンパク質予測では、G タンパク質共 役型受容体やイオンチャネルなど創薬標的タンパク質に関連した考察ができます。局在予 測では、核移行やミトコンドリア局在なども予測結果から生物学的な機能を推定すること も可能です。本チュートリアルでは、標的タンパク質同定を目的に、モチーフ検索として InterPro、シグナルペプチド予測に SignalP、膜タンパク質予測に TMHMM、局在予測に PSORT を用いて実習を行います。 【操作1】InterPro を用いたモチーフ検索 InterPro の Web サイトへアクセスします。http://www.ebi.ac.uk/interpro/
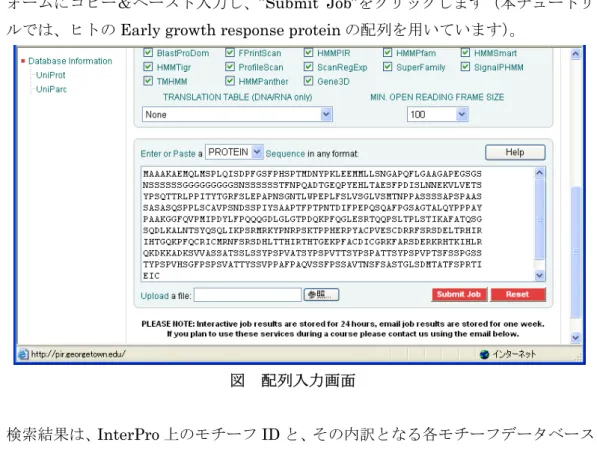
問い合わせ配列を入力するページに移動します。”InterProScan”をクリックします。 図 InterPro トップページ 解析を行いたい配列データ(アミノ酸一文字表記の文字列)を InterPro の配列入力フゲノム情報処理工学・実習テキスト
ォームにコピー&ペースト入力し、”Submit Job”をクリックします(本チュートリア ルでは、ヒトのEarly growth response protein の配列を用いています)。
図 配列入力画面 検索結果は、InterPro 上のモチーフ ID と、その内訳となる各モチーフデータベースの 検索結果がID、配列上のモチーフ位置のグラフィックスおよびモチーフ名で表示され ます。 図 モチーフ検索結果表示画面 モチーフ箇所を示す場所にマウスポインタを合わせると残基番号が表示されます。ま たPD*****や PF*****といった各モチーフ ID をクリックするとそのモチーフの詳細情
ゲノム情報処理工学・実習テキスト 報を確認できます。 図 モチーフ詳細画面へのリンク 【操作2】SignalP を用いたシグナルペプチド予測 SignalP の Web サイトへアクセスします。
http://www.cbs.dtu.dk/services/SignalP/
トップページに配列入力フォームがありますので、そこに IntePro の時と同様に解析 したいタンパク質のアミノ酸配列をコピー&ペーストにて入力します(本チュートリ アルでは、ヒトのSecretogranin-5 の配列を用いています)。 図 SignalP のトップ画面 解析するタンパク質の生物種に応じて、Organism group のパラメータを選択します。 その他のパラメータは、特に変更する必要はございません。”Submit”ボタンをクリッ クして解析を実行します。 計算結果は、ニューラルネットワーク(SignalP-NN result)と HMM(SignalP-HMM result)のそれぞれの解析について、グラフィックス及びテキスト形式で出力されます。
ゲノム情報処理工学・実習テキスト
図 ニューラルネットワークによる解析結果
ニューラルネットワークでは、既知のシグナルペプチドを持つタンパク質のデータセ ットで学習されたネットワークに基づいて 3 つのスコアでシグナルペプチドの可能性 を判断します。C score、S score、Y score は、それぞれ、切断部位、シグナルペプチ ド領域、S score を考慮した切断部位のスコアを表しています。これらのスコアをまと めた結果がテキストとして出力されます。HMM の結果では、切断部位(Cleavage prob.)、N 末端領域(n-region prob.)、疎水的領域(h-region prob.)そして、疎水性 領域から切断部位までの領域(c-region prob.)になりやすい確率をそれぞれスコア化 しています。SignalP-NN と SignalP-HMM の両者が同じ予測結果を示すことが理想 となります。 【操作3】PSORT による局在予測 PSORT の Web ページへアクセスします。
http://psort.hgc.jp/
解析したい配列の生物種によって、各 PSORT(WoLF PSORT, PSORT II, PSORT, iPSORT, PSORT-B)を選択します。本チュートリアルでは、グ Importin alpha-1 subunit を例に PSORTT II を使って解析を行います。
ゲノム情報処理工学・実習テキスト 図 PSORT バージョンの選択 解析したい配列(一文字表記)をアミノ酸配列入力フォームにコピー&ペーストで入 力し、”Submit”ボタンをクリックします。 図 配列情報の入力 PSORT では、複数の解析プログラムの解析結果よりシグナルペプチドや局在を予測し ます。Importin alpha-1 subunit の解析結果では、シグナルペプチドおよび膜貫通領域 を持たないことがわかります。また、局在予測については、ミトコンドリア局在シグ ナル解析等を考慮しながら、最終的な局在可能性を出力します。今回の解析では、細 胞質(cytoplasmic)および核(nuclear)への局在の可能性が高いと予測されています。
ゲノム情報処理工学・実習テキスト
ゲノム情報処理工学・実習テキスト
7.統合データベースを用いた標的タンパク質同定
【概 略】 標的タンパク質同定には、チュートリアル 2 で紹介した配列情報解析に基づいて同定す る他に、統合化データベースを用いた方法があります。近年の統合データベースは、配列-構造といったタンパク質の階層性のリンクだけではなく、関連したリガンド情報やパスウ ェイ情報まで網羅したデータベースとなっています。このような統合データベースには、 KEGG や DrugBank が有名です。特に DrugBank は、KEGG の内容も包括しており、ま たリガンドについても医薬品としての情報リソースまで関連付けがされていますので、創 薬標的タンパク質で利用しやくなっています。 【操作1】DrugBank を利用した化合物、遺伝子情報検索 DrugBank の Web サイトにアクセスします。http://redpoll.pharmacy.ualberta.ca/drugbank/
トップページにあるキーワード検索入力フォームを利用して検索を行います。キーワ ードにインフルエンザ治療薬の一つである”tamiflu”を入力し”Search”ボタンをクリッ クします。 図 DrugBank のトップページ 検索結果、一般名 Oseltamivir Phosphate として化合物がヒットします。詳細を確認 するために、DrugBank の Accession No である”APRD01148”をクリックします。ゲノム情報処理工学・実習テキスト
図 キーワードtamiflu の検索結果
詳細データでは、Oseltamivir Phosphate の構造式や KEGG や PubChem など他の化 合物データベースとのリンク、薬理活性等の情報が記載されています。
図 リガンド分子構造情報
化合物情報に続いて、Oseltamivir Phosphate が標的とするタンパク質の情報(Drug Target 1)が記載されています。化合物によって複数の標的タンパク質が存在する場合 には、Drug Target 2, 3…と続きます。ここでは、標的タンパク質の一次配列、機能(学 術的記載やGeneOntology 分類、Pfam モチーフなど)、DNA 配列、染色体位置などが 列挙されています。また標的タンパク質によっては、Pathway マップへのリンクも含 まれています。
ゲノム情報処理工学・実習テキスト
図 標的タンパク質情報
【操作2】Tamiflu をクエリーとした類似化合物検索
Tamiflu の検索結果のページの上部に”Show Similar Structure(s)”ボタンがあります。 これにより、化合物構造を基とした類似構造検索が可能です。検索対象は、初期設定 では、”Approved Drug(認可済医薬品)”となっていますが、”Small Molecule Drugs”, “Biotech Drugs”, “Nutraceutical Drugs”, “Experimental Drugs”, “All”が選択可能と なっています。本チュートリアルでは、初期設定のまま検索を実行します。”Show Similar Structure(s)”ボタンをクリックします。
図 類似化合物の検索
検索の結果、9 個の類似化合物がヒットし類似性スコアによってソートされています (この結果は、データベースの更新に伴い、変化する可能性があります)。
ゲノム情報処理工学・実習テキスト 図 Tamiflu の類似構造検索結果 それぞれのヒットの詳細は、”ACCESSION CODE”カラムの”DRUGCARD”ボタンを クリックすると確認できます。 【操作2】化合物構造による検索 操作1では、”tamiflu”という物質名称で検索を行いましたが、DrugBank では、ブラ ウザ上で化合物の構造式を直接スケッチして検索することができます。メニュー項目 の”ChemQuery”をクリックします。 図 ChemQuery メニューの選択 ChemQuery では、スケッチツールを使って分子構造を構築します。右のパレットから 原子を選択し、マウスの右ボタンを使って描画します。結合次数は、結合線をクリッ クすると単結合から変更可能な結合まで簡単に修正できます。上のパレットの左から 三番目にある Template ボタンをクリックすると環状構造など頻繁に利用される部分 構造が用意されています。
ゲノム情報処理工学・実習テキスト
図 スケッチ機能による問い合わせ化合物の構築
分子構造のスケッチが終了しましたら、”CLICK TO CONVERT TO MOL FILE”ボタ ンをクリックし、続いて、”CLICK TO SUBMIT QUERY”をクリックすると検索が実 行します。 【操作3】標的タンパク質の配列情報に基づく検索 操作1、2では、化合物情報に基づいた検索を行いました。DrugBank では、標的タ ンパク質の配列の相同性を用いてDrguBank にエントリーされている標的タンパク質 と関連化合物の情報を検索することができます。メニュー項目の”SeqSearch”をクリッ クします。ページには、アミノ酸配列入力フォームがありますので、検索したい配列 をコピー&ペーストで入力します。相同性検索にはBLAST が用いられます。 図 DrugBank の BLAST 検索ページ
ゲノム情報処理工学・実習テキスト
検索結果は、通常の BLAST 出力と同等ですが、各ヒットタンパク質に DrugBank の Accession No がついていますので、その Accession No をクリックすると詳細情報を確 認できます。
ゲノム情報処理工学・実習テキスト
8.タンパク質立体構造に基づく機能推定
【概 略】 構造ゲノミクスの進展によって、配列と立体構造がわかっているが、機能が未知のタン パク質に遭遇することが増えてきています。タンパク質の表面形状は、機能を予測する最 も有用な情報の一つになります。CASTp は、タンパク質立体構造データ(PDB 形式)を入 力情報に、活性ポケット候補部位を検索し、ポケットの体積や周辺残基の情報をグラフィ ックスを使いながら解析できる Web サイトです。CASTp などによる活性ポケット候補探 索は、創薬におけるドッキング計算のドッキング部位入力情報としても活用できます。ま た活性ポケットの体積情報を利用した化合物サイズの簡単なスクリーニング処理にも応用 できます。 【操作1】CASTp を用いた活性ポケット候補部位探索 CASTp の Web ページへアクセスします。http://sts.bioengr.uic.edu/castp/
CASTp では、PDB-ID(4 文字)で検索するのが一般的な利用方法となります。トッ プページに PDB-ID を入力する Query フォームがあります。本チュートリアルでは、 HIV プロテアーゼを代表して、”1hte”という PDB-ID を持つタンパク質を入力情報と します。Query フォームに 1hte を入力後、”Search”ボタンをクリックします。図 CASTp のトップページ
ゲノム情報処理工学・実習テキスト イルに記載されているアノテーション情報、下側の配列ウィンドウで構成されていま す。 図 CASTp 解析ページ 候補ポケットの観察には、表面積(Area)や体積(Vol)を参考に興味のあるポケット ID のチェックボックスにチェックを入れます。その結果、ポケットの該当箇所が中央 部のタンパク質立体構造のグラフィックスにアノテーションされます。同時に左下の エリアに選択したポケット周辺に存在する残基リストが表示されます。この残基リス トは、配列ウィンドウのアノテーションと同調しています。 図 候補ポケットの表示 【操作2】自前のタンパク質構造を入力としたCASTp解析方法 操作1のように解析対象のタンパク質が PDB に登録されていない場合(たとえば、モ デリングによりタンパク質立体構造を予測した結果をPDB ファイル形式で保有してい
ゲノム情報処理工学・実習テキスト
る場合)でもCASTp では、データファイルをサーバー上で Upload して解析を行うこ とができます。トップページの左側のメニューから”Calculation Request”を選択しま す。
図 Calculation Request ページへのリンク
Calculation Request でも PDB-ID を指定することが出来ます。その下にファイルを Upload する項目あります。解析したい立体構造データを PC などに用意して、参照ボ タンでPC 内のデータを選択します。指定後、Submit ボタンでポケット候補探索が実 行されます。結果は、操作1と同様な形式で出力されます。
ゲノム情報処理工学・実習テキスト
9.タンパク質立体構造に基づく酵素活性部位予測
【概 略】 タンパク質の機能には、配列の相同性や構造のフォールドが異なるが共通した酵素活性 機能を有しているものがあります。このような場合、配列相同性や配列情報に基づくモチ ーフ、立体構造予測だけでは機能予測が困難です。一方で、酵素のように酵素活性部位の 構造について研究が進んでいる分野では、細かな活性部位に関する情報のデータベース化 が進んでおります。Catalytic Site Atlas は、酵素活性部位に関するデータベースと解析ツ ールが備わったWeb サイトです。特にタンパク質立体構造情報を入力情報として与えれば、 JESS と呼ばれる酵素活性部位テンプレートの検索を行うことができます。これにより、全 体の折りたたみ構造や全体の配列相同性に依存なく活性部位を予測することができます。 Catalytic Site Atlas は、CASTp と同様、タンパク質立体構造情報に基づく機能予測手法の 一つとして広く利用されています。【操作1】Catalytic Site Atlas を用いたタンパク質立体構造に基づく酵素活性部位検索 Catalytic Site Atlas の Web ページへアクセスします。
http://www.ebi.ac.uk/thornton-srv/databases/CSA/
トップページからさらに“webserver”をクリックして解析のページに移動します。
図 Catalytic Site Atlas のトップページ
【操作2】データの読み込みと解析の実行
Catalytic Site Atlas では、既知の PDB に対して PDB-ID 等を検索情報に酵素活性部 位を検索するモードと、PDB に登録されていないタンパク質立体構造データに対して、
ゲノム情報処理工学・実習テキスト
既知の酵素活性部位テンプレートを用いて類似部位がないかを検索するモードの2つ があります。前者の場合にはPDB-ID を、後者の場合には、参照ボタンより自前の PC にある立体構造データ(PDB 形式)をアップロードし、”Submit Settings”ボタンをク リックして検索を実行します。検索の条件には、”Alpha and Beta carbon templates” と”Functional atom templates”があります。前者の方は、アミノ酸側鎖のα位および β位の炭素原子座標に基づいて行いますので、入力構造が予測構造や、側鎖配座の精 度があまり高くない場合に選択します。後者は、酵素活性に関与する原子の座標に基 づくものです。
図 解析データのアップロード
データが無事 Upload されたことが確認できるページに変わります。続いて、酵素活性 部位テンプレート検索プログラムJESS を実行します。”Run JESS”をクリックします。
図 テンプレート検索プログラムJESS の実行
ゲノム情報処理工学・実習テキスト
解析結果は、Summary テーブルで出力されます。例えば図の場合、tRNA synthetase (PDB-ID: 1qtq)の持つ酵素活性部位に類似した部分構造が入力データ中に見つかっ たことを示しています。ヒットした結果は、その結果の統計的有意性(E-value)によ って順位付けされています。酵素活性部位のような部分構造の検索では、実際の活性 部位とは関係のないランダムに起こりうるヒット(負の答え)との識別が非常に重要 となります。よって、E-value の値には注意しておきましょう。また、この値は Assessment 項目に反映されています。”Highly probable”や”Possible”を信頼できる結 果と考えてよいでしょう。
図 テンプレート検索結果
Detail をクリックするとヒットした酵素活性部位の Template 残基と対応する残基を 知ることができます。
図 ヒットした活性部位の詳細情報
ゲノム情報処理工学・実習テキスト
クすると酵素機能の情報や、このテンプレートを有している主なタンパク質の情報を 知ることができます。ページの後半部分では、酵素活性部位テンプレートの座標値と 配列相同性の関係や、感度、予測精度等のデータも表示されています。